Кинемаг - Kinemage

Кинемаг (сокращение от кинетический образ) представляет собой интерактивную графическую научную иллюстрацию. Часто используется для визуализации молекулы, особенно белки хотя он также может представлять другие типы трехмерных данных (например, геометрические фигуры, социальные сети,[1] или тетраэдры Базовый состав РНК ). Система kinemage разработана для оптимизации простоты использования, интерактивной производительности, а также восприятия и передачи подробной трехмерной информации. Информация о кинемаге хранится в текстовом файле, доступном для чтения человеком и машиной, который описывает иерархию экранных объектов и их свойств и включает необязательный пояснительный текст. Формат кинемага - это определенный химический тип MIME из 'chemical / x-kinemage' с расширением файла '.kin'.
Ранняя история
Кинемаги были впервые разработаны Дэвидом Ричардсоном в Медицинский факультет Университета Дьюка, для журнала Protein Society Белковая наука премьера которого состоялась в январе 1992 года.[2] В течение первых 5 лет (1992–1996) каждый выпуск журнала Protein Science включал в себя приложение дискета интерактивного, кинемага 3D компьютерная графика для иллюстрации многих статей, а также программного обеспечения Mage (кросс-платформенный, свободный, Открытый исходный код ) отображать их;[3] Дополнительные материалы о кинемаге по-прежнему доступны на веб-сайте журнала. Маг и РасМол[4] были первым широко используемым макросоммолекулярная графика программы для поддержки интерактивного отображения на персональные компьютеры. Кинемаги используются для обучения,[5][6] и для приложений к учебникам,[7][8] индивидуальное исследование и анализ макромолекулярных структур.

Исследования используют
В последнее время, когда стало доступно гораздо большее количество других молекулярная графика Инструменты, презентационное использование кинемагов было вытеснено широким спектром исследовательских применений, наряду с новыми функциями отображения и разработкой программного обеспечения, которое производит выходные данные в формате кинемага из других типов молекулярных вычислений. Всеатомный контактный анализ[9] добавляет и оптимизирует явные атомы водорода,[10] а затем использует участки точечной поверхности для отображения водородная связь, ван дер Ваальс, и стерический столкновение взаимодействия между атомами. Результаты можно использовать визуально (в кинемагах) и количественно для анализа детальных взаимодействий между молекулярными поверхностями,[11][12] наиболее широко с целью проверки и улучшения молекулярных моделей из экспериментальных рентгеновская кристаллография данные.[13][14][15][16] И Mage, и KiNG (см. Ниже) были улучшены для отображения данных кинематографом в более чем трех измерениях (перемещение между видами в различных трехмерных проекциях, раскрашивание и выбор потенциальных кластеров точек данных и переключение на параллельные координаты представление), используемый, например, для определения кластеров благоприятных конформаций остова РНК в 7-мерном пространстве двугранных углов остова между одной рибозой и следующей.[17]
Интернет-использование
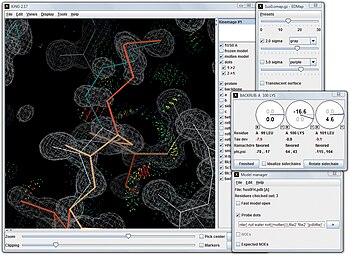
KiNG - программа просмотра кинемагов с открытым исходным кодом, написанная на языке программирования. Ява Ян Дэвис и Винсент Чен,[18] которые могут работать в интерактивном режиме либо автономно на пользовательском компьютере без сетевого подключения, либо как веб-сервис в страница в Интернете. Интерактивная природа кинемагов - их основная цель и атрибут. Чтобы оценить их природу, демонстрация KiNG в браузере содержит два примера, которые можно перемещать в 3D, а также инструкции по встраиванию кинемага на веб-страницу.[19] На рисунке ниже показано, что KiNG используется для ремоделирования боковой цепи лизина в кристаллической структуре с высоким разрешением. KiNG является одним из средств просмотра, представленных на каждой странице структуры сайта Protein Data Bank,[20] и отображает результаты проверки в 3D на сайте MolProbity.[21][22][23] Кинемаги также могут отображаться в иммерсивном виртуальная реальность систем с открытым исходным кодом KinImmerse.[24] Все программное обеспечение для отображения kinemage и контактов all-atom доступно бесплатно и с открытым исходным кодом на сайт кинемаге.
Смотрите также
- Молекулярная графика
- Ленточная диаграмма
- Сравнение программного обеспечения для моделирования молекулярной механики
Рекомендации
- ^ Freeman, L.C .; иерархия; и другие. (1998). «Изучение социальной структуры с помощью динамических трехмерных цветных изображений» (PDF). Социальные сети. 20 (2): 109–118. Дои:10.1016 / S0378-8733 (97) 00016-6.
- ^ Ричардсон, Д.; J.S. Ричардсон (январь 1992 г.). «Кинемаг: инструмент научного общения». Белковая наука. 1 (1): 3–9. Дои:10.1002 / pro.5560010102. ЧВК 2142077. PMID 1304880.
- ^ Нейрат, Х. (1992). "От редакции. Кинемаг: инструмент для научной иллюстрации". Белковая наука. 5 (11): 2147. Дои:10.1002 / pro.5560051101. ЧВК 2143300.
- ^ Сэйл Р. (1992). Материалы 10-й конференции Eurographics UK 1992 г.. Abingdon Press, Йорк.
- ^ Richardson, D.C .; J.S. Ричардсон (1994). «Кинемаги - простая макромолекулярная графика для интерактивного обучения и публикации». Тенденции в биохимических науках. 19 (3): 135–138. Дои:10.1016/0968-0004(94)90207-0. PMID 8203021.
- ^ Richardson, D.C .; J.S. Ричардсон (2002). «Обучение молекулярной 3-D грамотности». Биохимия и молекулярная биология образование. 30: 21–26. Дои:10.1002 / bmb.2002.494030010005.
- ^ Voet, D .; J. G. Voet; К. В. Пратт (1999). Основы биохимии. John Wiley & Sons, Нью-Йорк.
- ^ Branden, C.-I .; Дж. Туз (1999). Введение в структуру белка (2-е изд.). Garland Publishing, Inc., Нью-Йорк.
- ^ Word, J.M .; и другие. (1999). «Визуализация и количественная оценка молекулярного согласия: контактные точки малого зонда с явными атомами водорода». Журнал молекулярной биологии. 285 (4): 1711–1733. CiteSeerX 10.1.1.119.6173. Дои:10.1006 / jmbi.1998.2400. PMID 9917407.
- ^ Word, J.M .; и другие. (1999). «Аспарагин и глутамин: использование контактов атома водорода при выборе ориентации амида боковой цепи». Журнал молекулярной биологии. 285 (4): 1735–1747. CiteSeerX 10.1.1.323.6971. Дои:10.1006 / jmbi.1998.2401. PMID 9917408.
- ^ Word, J.M .; и другие. (2000). «Изучение стерических ограничений на мутации белков с помощью MAGE / PROBE». Белковая наука. 9 (11): 2251–2259. Дои:10.1110 / пс 9.11.2251. ЧВК 2144501. PMID 11152136.
- ^ Ричардсон, Дж. С.; Ричардсон, округ Колумбия (2002). «Натуральные белки β-листа используют негативный дизайн, чтобы избежать агрегации от края до края». Proc. Natl. Акад. Sci. Соединенные Штаты Америки. 99 (5): 2754–2759. Bibcode:2002PNAS ... 99.2754R. Дои:10.1073 / pnas.052706099. ЧВК 122420. PMID 11880627.
- ^ Richardson, D.C .; Ричардсон, Дж. С. (2001). «МАГ, ЗОНД и кинемаги». Международные таблицы для кристаллографии. F, глава 25.2.8: 727–730.
- ^ Ричардсон, Джейн С .; и другие. (2003). Новые инструменты и данные для улучшения структур с использованием All-Atom контактов. Методы в энзимологии: кристаллография макромолекул, часть D. Методы в энзимологии. 374. С. 385–412. Дои:10.1016 / S0076-6879 (03) 74018-X. ISBN 978-0-12-182777-9. PMID 14696383.
- ^ Хигман, В.А. ..; и другие. (2004). «Конформации боковых цепей аспарагина и глутамина в растворе и кристалле: сравнение лизоцима куриного яичного белка с использованием остаточных диполярных связей». Журнал биомолекулярного ЯМР. 30 (3): 327–346. Дои:10.1007 / s10858-004-3218-у. PMID 15754058.
- ^ Arendall III, W. B .; и другие. (2005). «Тест повышения точности модели в высокопроизводительной кристаллографии». Журнал структурной и функциональной геномики. 6 (1): 1–11. Дои:10.1007 / s10969-005-3138-4. PMID 15965733.
- ^ Richardson, J. S .; и другие. (2008). «Основа РНК: согласованные многоугольные конформеры и номенклатура модульных строк (вклад Консорциума по онтологии РНК)». РНК. 14 (3): 465–481. Дои:10.1261 / rna.657708. ЧВК 2248255. PMID 18192612.
- ^ Chen, V.B .; и другие. (2009). «KING (Kinemage, Next Generation): универсальная интерактивная программа молекулярной и научной визуализации». Белковая наука. 18 (11): 2403–2409. Дои:10.1002 / pro.250. ЧВК 2788294. PMID 19768809.
- ^ KiNG в браузере
- ^ «Банк данных белков». Архивировано из оригинал на 2008-08-28. Получено 2016-12-07.
- ^ MolProbity
- ^ Дэвис, И. В .; и другие. (2007). «MolProbity: всеатомные контакты и проверка структуры белков и нуклеиновых кислот». Исследования нуклеиновых кислот. 35 (Проблема с веб-сервером): W375 – W383. Дои:10.1093 / нар / гкм216. ЧВК 1933162. PMID 17452350.
- ^ Chen, V. B .; и другие. (2010). «MolProbity: проверка структуры всех атомов для кристаллографии макромолекул». Acta Crystallographica. D 66 (Пт 1): 12–21. Дои:10.1107 / S0907444909042073. ЧВК 2803126. PMID 20057044.
- ^ Block, J. N .; и другие. (2009). «KinImmerse: макромолекулярная VR для ансамблей ЯМР». Исходный код для биологии и медицины. 4: 3. Дои:10.1186/1751-0473-4-3. ЧВК 2650690. PMID 19222844.
внешняя ссылка
- Официальный веб-сайт, Оригинал Университета Дьюка, с примерами и программным обеспечением
- KiNG в браузере с интерактивными примерами
- Банк данных белков RCSB
- MolProbity: проверка структуры с помощью онлайн-кинемагов KiNG
