Теория расщепленного гена - Split gene theory
Эта статья поднимает множество проблем. Пожалуйста помоги Улучши это или обсудите эти вопросы на страница обсуждения. (Узнайте, как и когда удалить эти сообщения-шаблоны) (Узнайте, как и когда удалить этот шаблон сообщения)
|
В теория "расщепленного гена" к Periannan Senapathy это теория происхождения интроны, длинные некодирующие последовательности в генах эукариот, которые вмешиваются в экзоны.[1][2][3] Теория утверждает, что случайность первичных последовательностей ДНК допускает только небольшие (<600 пар оснований) открытые рамки для чтения, и что важные структуры интронов и регуляторные последовательности происходят из стоп-кодоны. В этой структуре, ориентированной прежде всего на интроны, сплайсосомный аппарат и ядро эволюционировали из-за необходимости объединить эти ORF (теперь «экзоны») в более крупные белки, и что бактериальные гены без интронов являются менее наследственными, чем расщепленные гены эукариот.
Теория предлагает решения ключевых вопросов, касающихся расщепленных эукариотических генов, включая экзоны, интроны, сплайсинговые соединения, точки ветвления и всю архитектуру расщепленных генов, на основе происхождения расщепленных генов из случайных генетических последовательностей. Он также предлагает возможные решения проблемы происхождения сплайсосомного аппарата, ядерной границы и эукариотической клетки. Эта теория привела к Алгоритм Шапиро-Сенапатии, который предоставляет методологию обнаружения сайтов сплайсинга, экзонов и расщепленных генов в эукариотической ДНК и является основным методом обнаружения мутаций сайтов сплайсинга в генах, вызывающих сотни заболеваний у тысяч пациентов во всем мире.
Подробности того, как была сформулирована теория расщепленного гена, и как эта теория подтверждается опубликованной литературой во всех аспектах генетических элементов эукариотического гена, представлены ниже.
Теория расщепленного гена требует отдельного происхождения всех видов эукариот. Это также требует, чтобы более простые прокариоты произошли от эукариот. Это полностью противоречит научному консенсусу об образовании эукариотических клеток путем эндосимбиоза бактерий. В 1994 году Сенапати написал книгу об этом аспекте своей теории - Самостоятельное рождение организмов. Он предположил, что все геномы эукариот были сформированы отдельно в первичном пуле. Голландский биолог Герт Кортофф раскритиковал теорию, поставив различные проблемы, которые нельзя объяснить теорией независимого происхождения. Он также указал, что различные эукариоты нуждаются в родительской заботе, и назвал это «проблемой загрузки». Не могло быть ни одного предка, нуждающегося в родительской заботе. Кортофф отмечает, что большая часть эукариот - паразиты. Теория сенапатии требует совпадения, чтобы объяснить их существование. [1] [2] Теория сенапатии также не может объяснить сильные доказательства общего происхождения (например, гомология, универсальный генетический код, эмбриология, летопись окаменелостей).[4]
Фон

Гены всех организмов, кроме бактерий, состоят из коротких участков, кодирующих белок (экзоны ) прерывается длинными последовательностями, которые вмешиваются в кодирующие последовательности (интроны ).[1][2] Когда ген экспрессируется, его последовательность ДНК копируется ферментом в последовательность «первичной РНК». РНК-полимераза. Затем «сплайсосомный» аппарат физически удаляет интроны из РНК-копии гена в процессе сплайсинга, оставляя только непрерывно связанную серию экзонов, которая становится «информационной» РНК (мРНК). Эта мРНК теперь «считывается» другим клеточным механизмом, называемым «рибосома, ”Для производства кодируемого белка. Таким образом, хотя интроны физически не удаляются из гена, последовательность гена читается так, как будто интронов никогда не существовало.
Экзоны обычно очень короткие, с размером ок. средняя длина около 120 оснований (например, в генах человека). Длина интронов широко варьируется от 10 до 500 000 оснований в геноме (например, в геноме человека), но длина экзонов имеет верхний предел около 600 оснований в большинстве эукариотических генов. Поскольку экзоны кодируют последовательности белков, они очень важны для клетки, но составляют только ~ 2% последовательностей генов. Интроны, напротив, составляют 98% последовательностей генов, но, по-видимому, имеют мало важных функций в генах, за исключением таких функций, как содержание энхансерных последовательностей и в редких случаях регуляторов развития.[5][6]
До того как Филип Шарп [7][8] из Массачусетского технологического института и Ричард Робертс [9] затем в Лаборатории Колд-Спринг-Харбор (в настоящее время в Биолабораториях Новой Англии) обнаружил интроны[10] в генах эукариот в 1977 году считалось, что кодирующая последовательность всех генов всегда находится на одном участке, ограниченном одной длинной открытой рамкой считывания (ORF). Открытие интронов стало большим сюрпризом для ученых, и тут же возник вопрос о том, как, почему и когда интроны попали в гены эукариот.
Вскоре стало очевидно, что типичный эукариотический ген во многих местах прерывается интронами, деля кодирующую последовательность на множество коротких экзонов. Также удивительным было то, что интроны были очень длинными, даже в сотни тысяч оснований (см. Таблицу ниже). Эти открытия также вызвали вопросы о том, почему много интронов встречается в гене (например, ~ 312 интронов встречается в TTN человеческого гена), почему они очень длинные и почему экзоны очень короткие.
| Символ гена | Длина гена (базы) | Самая длинная длина интрона (базы) | Количество интроны в гене |
|---|---|---|---|
| ROBO2 | 1,743,269 | 1,160,411 | 104 |
| KCNIP4 | 1,220,183 | 1,097,903 | 76 |
| ASIC2 | 1,161,877 | 1,043,911 | 18 |
| NRG1 | 1,128,573 | 956,398 | 177 |
| DPP10 | 1,403,453 | 866,399 | 142 |
| Самые длинные интроны в генах человека. | |||
Также было обнаружено, что сплайсосомный аппарат был очень большим и сложным с ~ 300 белками и несколькими молекулами SnRNA. Итак, вопросы также распространяются на происхождение сплайсосомы. Вскоре после открытия интронов стало очевидно, что соединения между экзонами и интронами с обеих сторон демонстрируют специфические последовательности, которые сигнализируют аппарату сплайсосомы о точном положении основания для сплайсинга. Как и почему возникли эти сигналы сращивания стыков, было еще одним важным вопросом, на который нужно было ответить.
Ранние предположения
Поразительное открытие интронов и расщепленной генной архитектуры эукариотических генов было драматичным и положило начало новой эре эукариотической биологии. Вопрос о том, почему эукариотические гены имеют целостную архитектуру, почти сразу же вызвал в литературе спекуляции и дискуссии.
Форд Дулиттл из Университета Далхаузи опубликовал в 1978 году статью, в которой выразил свои взгляды.[11] Он заявил, что большинство молекулярных биологов предполагали, что геном эукариот произошел из «более простого» и «примитивного» прокариотического генома, скорее похожего на геном кишечная палочка. Однако этот тип эволюции потребует, чтобы интроны были введены в непрерывные кодирующие последовательности бактериальных генов. Относительно этого требования Дулиттл сказал: «Чрезвычайно трудно представить, как информативно нерелевантные последовательности могут быть введены в уже существующие структурные гены без вредных эффектов». Он заявил: «Я хотел бы утверждать, что геном эукариот, по крайней мере в том аспекте его структуры, который проявляется как« гены по частям », на самом деле является примитивной исходной формой».
Джеймс Дарнелл из Университета Рокфеллера также выразили аналогичные взгляды в 1978 году.[12] Он заявил: «Различия в биохимии образования матричной РНК у эукариот по сравнению с прокариоты настолько глубоки, что предполагают, что последовательная эволюция прокариотических клеток в эукариотические клетки кажется маловероятной. Недавно обнаруженные несмежные последовательности в эукариотической ДНК, которые кодируют информационную РНК, могут отражать древнее, а не новое распределение информации в ДНК и то, что эукариоты развивались независимо от прокариот ».
Однако в явной попытке примириться с идеей о том, что РНК предшествовала ДНК в эволюции, и с концепцией трех эволюционных линий архея, бактерии и эукарии, и Дулиттл, и Дарнелл отклонились от своих первоначальных предположений в статье, опубликованной вместе в 1985 году.[13] Они предположили, что предок всех трех групп организмов, «прогенот, ’Имела структуру« гены в частях », из которой произошли все три линии. Они предположили, что на доклеточной стадии присутствуют примитивные гены РНК, которые имеют интроны, которые обратно транскрибируются в ДНК и образуют прогенот. Бактерии и археи произошли от прогенота, потеряв интроны, а «уркариот» развился из него, сохранив интроны. Позже эукариот произошел от уркариота, образовав ядро и получив митохондрии от бактерий. Затем многоклеточные организмы произошли от эукариот.
Эти авторы смогли предсказать, что различия между прокариотами и эукариотами были настолько глубокими, что эволюция от прокариотов к эукариотам была несостоятельной, и что оба они имели разное происхождение. Однако, помимо предположений о том, что гены доклеточной РНК должны иметь интроны, они не касались ключевых вопросов о том, откуда, как и почему интроны могли возникать в этих генах или какова их материальная основа. Не было никаких объяснений того, почему экзоны короткие, а интроны длинные, как возникают сплайсинговые соединения, что означают структура и последовательность сплайсинговых соединений и почему эукариотические геномы такие большие.
Примерно в то же время, когда Дулиттл и Дарнелл предположили, что интроны в эукариотических генах могут быть древними, Колин Блейк[14] из Оксфордского университета и Уолтер Гилберт[15][16] из Гарвардского университета (который получил Нобелевскую премию за изобретение метода секвенирования ДНК вместе с Фредом Сэнгером) независимо опубликовали свои взгляды на происхождение интронов. По их мнению, интроны возникли как спейсерные последовательности, которые сделали возможной рекомбинацию и перетасовку экзонов, кодирующих различные функциональные домены, с целью развития новых генов. Таким образом, новые гены были собраны из модулей экзонов, которые кодировали функциональные домены, складывающиеся области или структурные элементы из ранее существовавших генов в геноме предкового организма, тем самым развивая гены с новыми функциями. Они не указали, как произошли экзоны, представляющие структурные мотивы белков, или интроны, которые не кодируют белки. Кроме того, даже по прошествии многих лет обширный анализ нескольких тысяч белков и генов показал, что только в очень редких случаях гены проявляют предполагаемый феномен перетасовки экзонов.[17][18] Более того, несколько молекулярных биологов подвергли сомнению предложение о перетасовке экзонов с чисто эволюционной точки зрения как по методологическим, так и по концептуальным причинам, и в конечном итоге эта теория не оправдалась.
Гипотеза
Примерно в то же время, когда были открыты интроны, Senapathy задавалась вопросом, как могли возникнуть сами гены. Он предположил, что для возникновения любого гена в среде пребиотической химии должны быть генетические последовательности (РНК или ДНК). Основной вопрос, который он задал, заключался в том, как последовательности, кодирующие белок, могли возникнуть из первичных последовательностей ДНК на начальном этапе развития самых первых клеток.
Чтобы ответить на этот вопрос, он сделал два основных предположения: (i) до того, как могла появиться самовоспроизводящаяся клетка, молекулы ДНК были синтезированы в первичном бульоне путем случайного добавления 4 нуклеотидов без помощи матриц и (ii) нуклеотид последовательности, кодирующие белки, были выбраны из этих ранее существовавших случайных последовательностей ДНК в первичном бульоне, а не путем конструирования из более коротких кодирующих последовательностей. Он также предположил, что кодоны, должно быть, были установлены до происхождения первых генов. Если первичная ДНК действительно содержала случайные нуклеотидные последовательности, он спросил: существует ли верхний предел длины кодирующей последовательности, и, если да, то играет ли этот предел решающую роль в формировании структурных особенностей генов в самом начале происхождение генов?
Его логика была следующей. Средняя длина белков в живых организмах, включая эукариотические и бактериальные организмы, составляла ~ 400 аминокислот. Однако как у эукариот, так и у бактерий существовали гораздо более длинные белки, даже длиннее, чем от 10 000 до ~ 30 000 аминокислот.[19] Таким образом, кодирующая последовательность из тысяч оснований существовала в одном участке бактериальных генов. В отличие от этого кодирующая последовательность эукариот существовала только в коротких сегментах экзонов размером ок. 120 оснований независимо от длины белка. Если длины ORF кодирующей последовательности в случайных последовательностях ДНК были такими же длинными, как и у бактериальных организмов, то возможно, что в случайной ДНК присутствовали непрерывно длинные кодирующие гены. Это не было известно, поскольку распределение длин ORF в случайной последовательности ДНК никогда ранее не изучалось.
Поскольку в компьютере можно было генерировать случайные последовательности ДНК, Сенапати думал, что может задавать эти вопросы и проводить свои эксперименты на компьютере. Более того, когда он начал изучать этот вопрос, в базе данных Национального фонда биомедицинских исследований (NBRF) в начале 1980-х было достаточно информации о последовательностях ДНК и белков.
Проверка гипотезы
Происхождение интронов и структура расщепленного гена

Senapathy сначала проанализировала распределение длин ORF в компьютерных случайных последовательностях ДНК. Удивительно, но это исследование показало, что на самом деле существует верхний предел около 200 кодонов (600 оснований) для длин ORF. Самая короткая ORF (длина нулевого основания) была наиболее частой. При увеличении длины ORF их частота логарифмически уменьшалась, достигая почти нуля примерно на 600 основаниях. Когда была построена вероятность длин ORF в случайной последовательности, это также показало, что вероятность увеличения длин ORF снижалась экспоненциально и снижалась максимум до 600 оснований. Из этого «отрицательного экспоненциального» распределения длин ORF было обнаружено, что большинство ORF были чрезвычайно короче, чем даже максимум в 600 оснований.

Это открытие было неожиданным, потому что кодирующая последовательность для белка средней длины 400 AA (с ~ 1200 оснований кодирующей последовательности) и более длинных белков из тысяч AA (требующих> 10 000 оснований кодирующей последовательности) не могла бы встречаться на отрезке случайным образом. последовательность. Если бы это было так, то типичный ген с непрерывной кодирующей последовательностью не мог бы происходить из случайной последовательности. Таким образом, единственный возможный способ, которым любой ген мог происходить из случайной последовательности, состоял в том, чтобы разделить кодирующую последовательность на более короткие сегменты и выбрать эти сегменты из коротких ORF, доступных в случайной последовательности, а не увеличивать длину ORF за счет исключения множества последовательно расположенных встречающиеся стоп-кодоны. Этот процесс выбора коротких сегментов кодирующих последовательностей из доступных ORF для создания длинной ORF привел бы к расщепленной структуре гена.
Если эта гипотеза верна, последовательности ДНК эукариот должны иметь доказательства этого. Когда Senapathy построила график распределения длин ORF в последовательностях эукариотической ДНК, график был удивительно похож на график для случайной последовательности ДНК. Этот график также представляет собой отрицательное экспоненциальное распределение, которое заканчивается максимум на 600 основаниях. Это открытие было удивительным, потому что экзоны из эукариотических генов также имели максимальную длину около 600 оснований,[1][20][3] что точно совпало с максимальной длиной ORF, наблюдаемой как в случайной последовательности ДНК, так и в последовательности ДНК эукариот.
Таким образом, расщепленные гены произошли из случайных последовательностей ДНК путем выбора лучшего из коротких кодирующих сегментов (экзонов) и соединения их в процессе сплайсинга. Последовательности промежуточных интронов были остатками случайных последовательностей и, таким образом, были предназначены для удаления с помощью сплайсосомы. Эти результаты показали, что расщепленные гены могли происходить из случайных последовательностей ДНК с экзонами и интронами, как они обнаруживаются в современных эукариотических организмах. Нобелевский лауреат Маршалл Ниренберг, который расшифровал кодоны, заявил, что эти открытия убедительно показали, что теория расщепленных генов для происхождения интронов и расщепленной структуры генов должна быть верной.[1] Новый ученый освещал эту публикацию в «Длинном объяснении интронов».[21]
Известный молекулярный биолог доктор Колин Блейк из Оксфордского университета, который в 1979 году предложил гипотезу Гилберта-Блейка относительно происхождения интронов (см. Выше), заявил в своей публикации 1987 года под названием «Белки, экзоны и молекулярная эволюция», что расщепление сенапатии Теория генов всесторонне объяснила происхождение структуры расщепленного гена. Кроме того, он заявил, что он объяснил несколько ключевых вопросов, включая происхождение механизма сращивания:[14]
«Недавняя работа Senapathy в применении к РНК всесторонне объясняет происхождение сегрегированной формы РНК на кодирующие и некодирующие области. Это также указывает на то, почему механизм сплайсинга был разработан в начале изначальной эволюции. Он обнаружил, что распределение длин рамок считывания в случайной нуклеотидной последовательности точно соответствует наблюдаемому распределению размеров экзонов эукариот. Они были ограничены областями, содержащими стоп-сигналы, сообщения о прекращении построения полипептидной цепи, и, таким образом, были некодирующими областями или интронами. Таким образом, наличие случайной последовательности было достаточным для создания у изначального предка сегрегированной формы РНК, наблюдаемой в структуре эукариотического гена. Более того, случайное распределение также показывает отсечку на уровне 600 нуклеотидов, что предполагает, что максимальный размер для раннего полипептида составлял 200 остатков, что опять же наблюдается в максимальном размере эукариотического экзона. Таким образом, в ответ на эволюционное давление, направленное на создание более крупных и сложных генов, фрагменты РНК были соединены вместе с помощью механизма сплайсинга, который удалял интроны. Следовательно, раннее существование как интронов, так и сплайсинга РНК у эукариот представляется весьма вероятным с простой статистической основы. Эти результаты также согласуются с линейной зависимостью, обнаруженной между количеством экзонов в гене для конкретного белка и длиной полипептидной цепи ».

Происхождение стыков
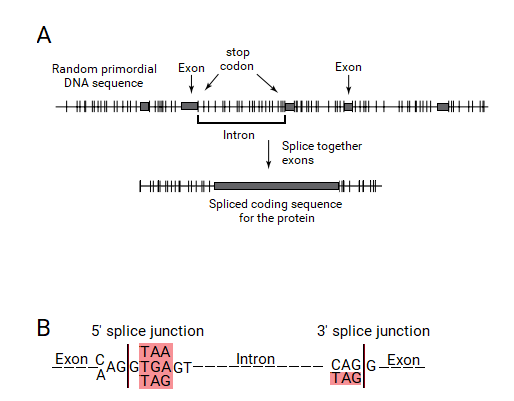
Согласно теории расщепленного гена, экзон определяется ORF. Это потребует создания механизма распознавания ORF. Поскольку ORF определяется непрерывно кодирующей последовательностью, ограниченной стоп-кодонами, эти концы стоп-кодонов должны распознаваться этой системой распознавания экзон-интрон. Эта система могла бы определять экзоны по присутствию стоп-кодона на концах ORF, который должен быть включен в концы интронов и устранен в процессе сплайсинга. Таким образом, интроны должны содержать стоп-кодон на своих концах, который должен быть частью последовательностей сплайсинговых соединений.
Если эта гипотеза верна, расщепленные гены современных живых организмов должны содержать стоп-кодоны точно на концах интронов. Когда Senapathy проверила эту гипотезу на сплайсинговых соединениях эукариотических генов, было удивительно, что подавляющее большинство сплайсинговых соединений действительно содержало стоп-кодон на концах интронов, прямо за пределами экзонов. Фактически, было обнаружено, что эти стоп-кодоны образуют «каноническую» последовательность сплайсинга GT: AG, причем три стоп-кодона встречаются как часть сильных консенсусных сигналов. Таким образом, основная теория расщепленных генов для происхождения интронов и структуры расщепленных генов привела к пониманию того, что сплайсинговые соединения происходят из стоп-кодонов.[2]
| Кодон | Количество вхождений в донорском сигнале | Количество вхождений в акцепторном сигнале |
|---|---|---|
| TAA | 370 | 0 |
| TGA | 293 | 0 |
| ТЕГ | 64 | 234 |
| CAG | 7 | 746 |
| Другой | 297* | 50 |
| Общий | 1030 | 1030 |
| Частота стоп-кодонов в донорных и акцепторных последовательностях сплайс-стыков.[2] Данные о последовательностях только около 1000 экзон-интронных переходов были доступны, когда * Более 70% составляют НАЛОГ [ТАТ = 75; ТАС = 59; TGT = 70]. | ||
Все три стоп-кодона (TGA, TAA и TAG) были обнаружены после одного основания (G) в начале интронов. Эти стоп-кодоны показаны в консенсусном каноническом донорском сплайсинге как AG: GT (A / G) GGT, где TAA и TGA являются стоп-кодонами, и дополнительный TAG также присутствует в этом положении. Помимо кодона CAG, на концах интронов был обнаружен только TAG, который является стоп-кодоном. Канонический акцепторный сплайсинг показан как (C / T) AG: GT, в котором TAG является стоп-кодоном. Эти консенсусные последовательности ясно показывают присутствие стоп-кодонов на концах интронов, граничащих с экзонами во всех эукариотических генах, таким образом обеспечивая сильное подтверждение теории расщепленного гена.Маршалл Ниренберг снова заявил, что эти наблюдения полностью подтверждают теорию расщепленных генов для происхождения последовательностей сплайс-соединений из стоп-кодонов, который был рецензентом этой статьи.[2] Новый ученый освещала эту публикацию в «Экзоны, интроны и эволюция».[22]
Вскоре после открытия интронов Доктора Филип Шарп и Ричард Робертс стало известно, что мутации внутри сплайсинговых соединений могут приводить к заболеваниям. Сенапатия показала, что мутации в основаниях стоп-кодонов (канонических основаниях) вызывают больше болезней, чем мутации в неканонических основаниях.[1]
Последовательность точки ветвления (лариата)
Промежуточным этапом в процессе сплайсинга эукариотической РНК является формирование лариатической структуры. Он закреплен на аденозин остаток в интроне между 10 и 50 нуклеотидами выше 3'-сайта сплайсинга. Короткая консервативная последовательность (последовательность точки ветвления) действует как сигнал распознавания для сайта образования лариата. В процессе сплайсинга эта консервативная последовательность ближе к концу интрона образует лариатную структуру с началом интрона.[23] Заключительный этап процесса сплайсинга происходит, когда два экзона соединяются и интрон высвобождается в виде лариатной РНК.[24]
Несколько исследователей обнаружили последовательности точек ветвления в разных организмы[23] включая дрожжи, человека, плодовую муху, крысу и растения. Сенапатия обнаружила, что во всех этих последовательностях точек ветвления кодон, заканчивающийся в точке ветвления аденозин последовательно является стоп-кодоном.Что интересно, два из трех стоп-кодонов (TAA и TGA) почти всегда встречаются в этой позиции.
| Организм | Последовательность Lariat Consensus |
|---|---|
| Дрожжи | TACTAAC |
| Гены бета-глобина человека | CTGAC CTAAТ CTGAТ CTAAC CTCAC |
| Дрозофила | CTAAТ |
| Крысы | CTGAC |
| Растения | (C / T)Т (А / Г) А(Т / К) |
| Постоянное присутствие стоп-кодонов в сигнальных последовательностях точки ветвления. Последовательности лариата (точки ветвления) были идентифицированы из множества различных организмы.Эти последовательности последовательно показывают, что кодон, заканчивающийся на разветвление аденозин - это стоп-кодон, TAA или TGA, которые показаны красным. | |
Эти открытия привели к тому, что Senapathy предположил, что сигнал точки ветвления происходит от стоп-кодонов. Обнаружение того, что два разных стоп-кодона (TAA и TGA) встречаются в сигнале лариата с точкой ветвления в качестве третьего основания стоп-кодонов, подтверждает это предположение. Поскольку точка ветвления лариата происходит на последнем аденине стоп-кодона, возможно, что сплайсосомный аппарат, возникший для устранения многочисленных стоп-кодонов из первичной последовательности РНК, создал сигнал вспомогательной стоп-кодоновой последовательности в качестве последовательность лариат, чтобы помочь его функции сращивания.[2]
Считается, что малая ядерная РНК U2, обнаруженная в комплексах сплайсинга, способствует сплайсингу, взаимодействуя с последовательностью лариата.[25] Комплементарные последовательности как для последовательности лариата, так и для акцепторного сигнала присутствуют в сегменте всего из 15 нуклеотидов в РНК U2. Кроме того, РНК U1 была предложена в качестве ориентира при сплайсинге для идентификации точного соединения сплайсинга донора посредством комплементарного спаривания оснований. Таким образом, консервативные области РНК U1 включают последовательности, комплементарные стоп-кодонам. Эти наблюдения позволили Senapathy предсказать, что стоп-кодоны действовали в качестве источника не только сигналов сплайс-соединения и сигнала лариата, но также и некоторых малых ядерных РНК.
Регуляторные последовательности генов
Доктор Сенапати также предположил, что последовательности, регулирующие экспрессию генов (последовательности промотора и сайта присоединения поли-A), также могли происходить из стоп-кодонов. Консервативная последовательность AATAAA существует почти в каждом гене на небольшом расстоянии ниже конца сообщения, кодирующего белок, и служит сигналом для добавления поли (A) в копию мРНК гена.[26] Этот сигнал последовательности поли (А) содержит стоп-кодон, ТАА. Последовательность, расположенная вскоре после этого сигнала, которая считается частью полного сигнала поли (A), также содержит стоп-кодоны TAG и TGA.
Эукариотическая РНК-полимераза-II-зависимая промоутеры может содержать ТАТА-бокс (консенсусную последовательность ТАТААА), который содержит стоп-кодон ТАА. Бактериальные промоторные элементы на -10 оснований демонстрируют TATA-бокс с консенсусом TATAAT (который содержит стоп-кодон TAA), а на -35 основаниях демонстрирует консенсус TTGACA (содержащий стоп-кодон TGA). Таким образом, на эволюцию всего механизма процессинга РНК, по-видимому, повлияло слишком частое появление стоп-кодонов в последовательности ДНК, что сделало стоп-кодоны фокусными точками для процессинга РНК.
Стоп-кодоны являются ключевыми частями каждого генетического элемента эукариотического гена.

| Генетический элемент | Консенсусная последовательность |
|---|---|
| Промоутер | TATAAТ |
| Последовательность сращивания донора | CAG: GTAAGT CAG: GTGAGT |
| Последовательность соединения акцептора | (З / Т) 9 ...ТЕГ: GT |
| Последовательность лариата | CTGAC CTAAC |
| Сайт добавления Поли-А | TATAAА |
| Последовательное появление стоп-кодонов в генетических элементах эукариотических генов.Консенсусные последовательности различных генетических элементов в эукариотических показаны гены. Стоп-кодон (ы) в каждой из этих последовательностей окрашен в красный цвет. | |
Доктора Сенапати работа, основанная на его теории расщепленного гена, раскрыла, что стоп-кодоны являются ключевыми частями в каждом генетическом элементе в эукариотические гены. Таблица и рисунок выше показывают, что ключевые части основных элементов промотора, сигнала лариата (точки ветвления), сигналов сплайсинга донора и акцептора и сигнала добавления поли-A состоят из одного или нескольких стоп-кодонов. Этот вывод является убедительным подтверждением теории расщепленных генов, согласно которой основной причиной парадигмы полного расщепления генов является происхождение расщепленных генов из случайных последовательностей ДНК, причем для их определения природа использовала случайное распределение чрезвычайно высокой частоты стоп-кодонов. генетические элементы.
Почему экзоны короткие, а интроны длинные?
Исследования, основанные на теории расщепленных генов, проливают свет на другие основные вопросы экзонов и интронов. Экзоны эукариоты обычно короткие (экзоны человека в среднем ~ 120 оснований и могут быть всего лишь 10 оснований), а интроны обычно очень длинные (в среднем ~ 3000 оснований и могут составлять несколько сотен тысяч оснований), например гены RBFOX1, CNTNAP2, PTPRD и DLG2. Senapathy предоставила правдоподобный ответ на эти вопросы, который до сих пор остается единственным объяснением. Основываясь на теории расщепленных генов, экзоны эукариотических генов, если они происходят из случайных последовательностей ДНК, должны соответствовать длинам ORF из случайной последовательности и, возможно, должны быть около 100 оснований (близко к средней длине ORF в случайной последовательности) . Последовательности генома живых организмов, например человека, имеют точно такую же среднюю длину в 120 оснований для экзонов и самые длинные экзоны из 600 оснований (за некоторыми исключениями), что соответствует длине самых длинных случайных ORF.[1][2][3][20]
Если бы расщепленные гены возникли из случайных последовательностей ДНК, то интроны были бы длинными по нескольким причинам. Стоп-кодоны располагаются в кластерах, что приводит к многочисленным последовательным очень коротким ORF, а более длинные ORF, которые можно определить как экзоны, будут более редкими. Кроме того, наилучшие параметры кодирующей последовательности для функциональных белков должны быть выбраны из длинных ORF в случайной последовательности, что может встречаться редко. Кроме того, комбинация донорных и акцепторных последовательностей сплайсинговых соединений в пределах коротких отрезков сегментов кодирующей последовательности, которые будут определять границы экзонов, будет редко встречаться в случайной последовательности. Эти комбинированные причины сделали бы интроны очень длинными по сравнению с длиной экзонов.
Почему эукариотические геномы большие?
Эта работа также объясняет, почему геномы очень большие, например, геном человека с тремя миллиардами оснований, и почему только очень небольшая часть генома человека (~ 2%) кодирует белки и другие регуляторные элементы.[27][28] Если бы расщепленные гены произошли из случайных первичных последовательностей ДНК, они бы содержали значительное количество ДНК, которая была бы представлена интронами. Кроме того, геном, собранный из случайной ДНК, содержащей расщепленные гены, также будет включать межгенную случайную ДНК. Таким образом, зарождающиеся геномы, происходящие из случайных последовательностей ДНК, должны были быть большими, независимо от сложности организма.
Наблюдение за тем, что геномы нескольких организмов, например лука (~ 16 миллиардов оснований)[29]) и саламандры (~ 32 млрд баз[30]) намного больше, чем у человека (~ 3 млрд оснований[31][32]), но организмы не сложнее, чем человек, что подтверждает эту теорию расщепленного гена. Кроме того, данные о том, что геномы некоторых организмов меньше, хотя они содержат по существу такое же количество генов, что и человеческий, например, C. elegans (размер генома ~ 100 миллионов оснований, ~ 19000 генов)[33] и Arabidopsis thaliana (размер генома ~ 125 миллионов оснований, ~ 25000 генов),[34] добавляет поддержку этой теории. Теория расщепленных генов предсказывает, что интроны в расщепленных генах в этих геномах могут быть «сокращенной» (или удаленной) формой по сравнению с более крупными генами с длинными интронами, что приводит к сокращению геномов.[1][20] Фактически, исследователи недавно предположили, что эти меньшие геномы на самом деле являются уменьшенными геномами, что добавляет поддержку теории расщепления генов.[35]
Происхождение сплайсосомного аппарата и эукариотического ядра
Исследования Senapathy также обращаются к происхождению сплайсосомного аппарата, который вырезает интроны из РНК-транскриптов генов. Если бы расщепленные гены произошли от случайной ДНК, то интроны стали бы ненужной, но неотъемлемой частью эукариотических генов вместе со сплайсинговыми соединениями на их концах. Сплайсосомный аппарат потребуется для их удаления и для того, чтобы короткие экзоны могли быть линейно сплайсированы вместе в виде непрерывно кодирующей мРНК, которая может транслироваться в полный белок. Таким образом, теория расщепленных генов показывает, что весь сплайсосомный аппарат возник из-за происхождения расщепленных генов из случайных последовательностей ДНК и удаления ненужных интронов.[1][2]
Как отмечалось выше, Колин Блейк, автор теории происхождения интронов и экзонов Гилберта-Блейка, заявляет: «Недавняя работа Senapathy в применении к РНК всесторонне объясняет происхождение сегрегированной формы РНК на кодирующие и некодирующие. регионы. Это также указывает на то, почему механизм сплайсинга был разработан в начале изначальной эволюции ».[14]
Сенапатия также предложила правдоподобное механистическое и функциональное объяснение возникновения эукариотического ядра, что является важным вопросом в биологии.[1][2] Если бы транскрипты расщепленных генов и сплайсированных мРНК присутствовали в клетке без ядра, рибосомы попытались бы связываться как с несращенным первичным РНК-транскриптом, так и с сплайсированной мРНК, что привело бы к молекулярному хаосу. Если граница возникла, чтобы отделить процесс сплайсинга РНК от трансляции мРНК, можно избежать этой проблемы молекулярного хаоса. Это именно то, что находится в эукариотических клетках, где сплайсинг первичного транскрипта РНК происходит внутри ядра, а сплайсированная мРНК транспортируется в цитоплазму, где рибосомы переводят их в белки. Граница ядра обеспечивает четкое разделение сплайсинга первичной РНК и трансляции мРНК.
Происхождение эукариотической клетки
Эти исследования, таким образом, привели к возможности того, что первичная ДНК с по существу случайной последовательностью дала начало сложной структуре расщепленных генов с экзонами, интронами и сплайсинговыми соединениями. Они также предсказывают, что клетки, несущие эти расщепленные гены, должны были быть комплексными с ядерной цитоплазматической границей и иметь сплайсосомный аппарат. Таким образом, вполне возможно, что самая ранняя клетка была сложной и эукариотической.[1][2][3][20] Удивительно, но результаты обширных сравнительных исследований геномики нескольких организмов за последние 15 лет убедительно показывают, что самые ранние организмы могли быть очень сложными и эукариотическими и могли содержать сложные белки,[36][37][38][39][40][41][42] точно так, как предсказывает теория сенапати.
Сплайсосома - это очень сложный механизм внутри эукариотической клетки, содержащий ~ 200 белков и несколько SnRNP. В своей статье [43] “Сложная сплайсосомная организация, унаследованная от современных эукариот», - заявляют молекулярные биологи Лесли Коллинз и Дэвид Пенни:« Мы начинаем с гипотезы о том, что ... сплайсосома усложнялась на протяжении всей эволюции эукариот. Однако изучение распределения сплайсосомных компонентов показывает, что сплайсосома не только присутствовала у эукариотического предка, но также содержала большинство ключевых компонентов, обнаруженных у современных эукариот. ... последний общий предок современных эукариот, кажется, демонстрирует большую часть молекулярной сложности, наблюдаемой сегодня ». Это говорит о том, что самые ранние эукариотические организмы были очень сложными и содержали сложные гены и белки, как и предсказывает теория расщепленных генов.
Происхождение бактериальных генов
Основываясь на теории расщепленных генов, только гены, расщепленные на короткие экзоны и длинные интроны с максимальной длиной экзона ~ 600 оснований, могли встречаться в случайных последовательностях ДНК. Гены с длинными непрерывными кодирующими последовательностями длиной в тысячи оснований и длиной от 10 000 до 90 000 оснований, которые встречаются во многих бактериальных организмах[19] были практически невозможны. Однако бактериальные гены могли возникнуть из расщепленных генов в результате потери интронов, что, по-видимому, является единственным способом получить длинные кодирующие последовательности. Это также лучший способ, чем увеличение длины ORF с очень коротких случайных ORF до очень длинных ORF путем специального удаления стоп-кодонов путем мутации.[1][2][3]
| Размер гена (основания) | Количество генов |
|---|---|
| 5,000 - 10,000 | 3,029 |
| 10,000 - 15,000 | 492 |
| 15,000 - 20,000 | 131 |
| 20,000 - 25,000 | 39 |
| >25,000 | 41 |
| Чрезвычайно длинные кодирующие последовательности встречаются в бактериальных генах как очень длинные ORF. Тысячи генов длиной более 5000 оснований кодируют белки, которые более 2000 аминокислот существуют во многих бактериальных геномах. Самые длинные гены составляют ~ 90 000 оснований, кодирующих белки длиной ~ 30 000 аминокислот. Каждый из них гены встречаются в единственном участке кодирующей последовательности (ORF) без прерывания кодоны или промежуточные интроны. Данные взяты из Думайте масштабно - гигантские гены в бактериях.[19] | |
Согласно теории расщепленного гена, этот процесс потери интрона мог произойти из-за пребиотической случайной ДНК. Эти непрерывно кодирующие гены могут быть плотно организованы в бактериальных геномах без каких-либо интронов и быть более упорядоченными. Согласно Senapathy, ядерная граница, необходимая для клетки, содержащей расщепленные гены в ее геноме (см. Происхождение ядра эукариотической клетки, выше) не потребуется для клетки, содержащей только непрерывно кодирующие гены. Таким образом, у бактериальных клеток не образовалось ядро. Основываясь на теории расщепленных генов, эукариотические геномы и бактериальные геномы могли независимо возникнуть из расщепленных генов в первичных случайных последовательностях ДНК.
Алгоритм Шапиро-Сенапатии
Основываясь на теории расщепленного гена, Senapathy разработала вычислительные алгоритмы для обнаружения донорных и акцепторных сайтов сплайсинга, экзонов и полного расщепленного гена в геномной последовательности. Он разработал метод матрицы весовых коэффициентов (PWM), основанный на частоте четырех оснований в согласованных последовательностях донора и акцептора в разных организмах, чтобы идентифицировать сайты сплайсинга в данной последовательности. Кроме того, он сформулировал первый алгоритм для поиска экзонов, основанный на требовании, чтобы экзоны содержали донорную последовательность (на 5'-конце) и акцепторную последовательность (на 3'-конце), а также ORF, в которой должен встречаться экзон. , и еще один алгоритм для поиска полного расщепленного гена. Эти алгоритмы все вместе известны как алгоритм Шапиро-Сенапатии (S&S).[44][45]
Этот Алгоритм Шапиро-Сенапатии помогает в выявлении мутаций сплайсинга, вызывающих многочисленные заболевания и побочные реакции на лекарства.[44][45] Используя алгоритм S&S, ученые определили мутации и гены, которые вызывают многочисленные виды рака, наследственные нарушения, заболевания иммунодефицита и неврологические расстройства (см. здесь подробнее). Он все чаще используется в клинической практике и исследованиях не только для поиска мутаций в известных болезнетворных генах у пациентов, но и для открытия новых генов, вызывающих различные заболевания. Кроме того, он используется для определения скрытых сайтов сплайсинга и определения механизмов, с помощью которых мутации в них могут влиять на нормальный сплайсинг и приводить к различным заболеваниям. Он также используется для решения различных вопросов фундаментальных исследований человека, животных и растений.
Широкое использование этого алгоритма в биологических исследованиях и клинических приложениях по всему миру добавляет доверия к теории расщепленного гена, поскольку этот алгоритм произошел от теории расщепленного гена. Выводы, основанные на S&S, затронули основные вопросы биологии эукариот и их приложений в медицине. Эти приложения могут расширяться за счет областей клинической геномики и фармакогеномика усилить свои исследования с помощью мегапроектов секвенирования, таких как проект All of Us[46] Это позволит секвенировать миллион человек, а в будущем - миллионы пациентов, участвующих в клинической практике и исследованиях.
Подтверждающие доказательства
Если теория расщепленных генов верна, можно ожидать, что структурные особенности расщепленных генов, предсказанные на основе компьютерно смоделированных случайных последовательностей, будут иметь место в реальных расщепленных генах эукариот. Это то, что мы находим в большинстве известных расщепленных генов у современных эукариот. Последовательности эукариот демонстрируют почти идеальное отрицательное экспоненциальное распределение длин ORF с верхним пределом 600 оснований (за редкими исключениями).[1][2][20][3] Кроме того, за редким исключением, экзоны эукариотических генов попадают в этот верхний максимум в 600 оснований.
Более того, если эта теория верна, экзоны должны быть ограничены стоп-кодонами, особенно на 3 ’концах экзонов (то есть на 5’ конце интронов). На самом деле, как и предполагалось, в большинстве известных генов они более точно разграничены на 3 ’концах экзонов и менее сильно на 5’ концах.[1][2][20][3] Эти стоп-кодоны являются наиболее важными функциональными частями обоих сплайсинговых соединений (канонические основания GT: AG). Таким образом, теория дает объяснение «консервативным» сплайсинговым соединениям на концах экзонов и потере этих стоп-кодонов вместе с интронами при их сплайсинге. Если эта теория верна, сплайсинговые соединения должны быть случайным образом распределены в последовательностях эукариотической ДНК, и так оно и есть.[3][23][44][45] Сплайсинговые соединения, присутствующие в генах транспортной РНК и генах рибосомной РНК, которые не кодируют белки и в которых стоп-кодоны не имеют функционального значения, не должны содержать стоп-кодоны, и это снова наблюдается. Сигнал лариата, другая последовательность, участвующая в процессе сплайсинга, также содержит стоп-кодоны.[1][2][3][20][23][44][45]
Если теория расщепленного гена верна, то интроны не должны кодировать. Это точно установлено для современных эукариотических организмов, даже когда интроны состоят из сотен тысяч оснований. Они также должны быть в основном нефункциональными, и они есть. За исключением некоторых последовательностей интронов, включая донорные и акцепторные сигнальные последовательности сплайсинга и последовательности точек ветвления, и, возможно, энхансеры сплайсинга интронов, которые встречаются на концах интронов, которые помогают в удалении интронов, подавляющее большинство интронов лишены каких-либо функций. . Теория расщепленного гена не исключает того, что редкие последовательности внутри интронов случайно демонстрируют функциональные элементы, которые могут быть использованы геномом и клеткой, особенно потому, что интроны очень длинные, что, как оказалось, верно. Все эти находки показывают, что предсказания теории расщепленных генов точно подтверждаются структурными и функциональными характеристиками основных генетических элементов расщепленных генов современных эукариотических организмов.
Если расщепленные гены произошли от случайных первичных последовательностей ДНК, как это предлагается в теории расщепленных генов, могло бы быть доказательство того, что они присутствовали в самых ранних организмах. Фактически, используя сравнительный анализ современных данных генома нескольких живых организмов, ученые обнаружили, что характеристики расщепленных генов, присутствующие у современных эукариот, восходят к самым ранним организмам, появившимся на Земле. Эти исследования показывают, что самые ранние организмы могли содержать богатые интронами расщепленные гены и сложные белки, которые встречаются в современных живых организмах.[47][48][49][50][51][52][53][54][55]
Кроме того, используя другой вычислительно-аналитический метод, известный как «анализ максимального правдоподобия», ученые обнаружили, что самые ранние эукариотические организмы должны были содержать те же гены, что и современные живые организмы, с даже более высокой плотностью интронов.[56] Кроме того, сравнительная геномика многих организмов, включая базальные эукариоты (которые считаются примитивными эукариотическими организмами, такими как Amoeboflagellata, Diplomonadida и Parabasalia) показали, что богатые интронами расщепленные гены, сопровождаемые полностью сформированной сплайсосомой из современных сложных организмов, присутствовали в самых ранних организмах, и что самые ранние организмы были чрезвычайно сложными с все компоненты эукариотической клетки.[57][47][58][59][60][56]
Эти литературные данные в точности совпадают с предсказаниями теории расщепленных генов, почти с математической точностью, обеспечивая замечательную поддержку. Эта теория подтверждается результатами сравнительного анализа фактических последовательностей эукариотических генов с последовательностями случайных последовательностей ДНК, сгенерированных компьютером. Кроме того, сравнительный анализ данных генома многих организмов, живущих сегодня, несколькими группами ученых показывает, что самые ранние организмы, появившиеся на Земле, имели богатые интронами расщепленные гены, кодирующие сложные белки и клеточные компоненты, такие как те, что обнаруживаются в современных эукариотических организмах. . Таким образом, теория расщепленного гена обеспечивает комплексные решения для всех структурных и функциональных особенностей архитектуры расщепленного гена с убедительными подтверждающими доказательствами из опубликованной литературы.
Избранные публикации
- Шапиро, Марвин Б .; Senapathy, Periannan (1987). «Соединения сплайсинга РНК различных классов эукариот: статистика последовательностей и функциональное значение в экспрессии генов». Исследования нуклеиновых кислот. 15 (17): 7155–7174. Дои:10.1093 / nar / 15.17.7155. ЧВК 306199. PMID 3658675.
- Senapathy, P. (1988). «Возможная эволюция сигналов сплайс-соединения в генах эукариот из стоп-кодонов». Proc Natl Acad Sci U S A. 85 (4): 1129–33. Bibcode:1988ПНАС ... 85.1129С. Дои:10.1073 / pnas.85.4.1129. ЧВК 279719. PMID 3422483.
- Senapathy, P; Шапиро, МБ; Харрис, Н.Л. (1990). Соединения сплайсинга, сайты точек ветвления и экзоны: статистика последовательностей, идентификация и приложения в геномном проекте. Методы в энзимологии. 183. стр.252–78. Дои:10.1016/0076-6879(90)83018-5. ISBN 9780121820848. PMID 2314278.
- Harris, N.L .; Сенапати, П. (1990). «Распределение и консенсус сигналов точки ветвления в эукариотических генах: компьютеризированный статистический анализ». Нуклеиновые кислоты Res. 18 (10): 3015–9. Дои:10.1093 / nar / 18.10.3015. ЧВК 330832. PMID 2349097.
- Сенапати, П. (1986). «Происхождение эукариотических интронов: гипотеза, основанная на статистике распределения кодонов в генах, и ее последствия». Proc Natl Acad Sci U S A. 83 (7): 2133–7. Bibcode:1986ПНАС ... 83.2133С. Дои:10.1073 / pnas.83.7.2133. ЧВК 323245. PMID 3457379.
- Regulapati, R .; Bhasi, A .; Singh, C.K .; Сенапати, П. (2008). «Происхождение расщепленной структуры сплайсосомных генов из случайных генетических последовательностей». PLoS ONE. 3 (10): 10. Bibcode:2008PLoSO ... 3.3456R. Дои:10.1371 / journal.pone.0003456. ЧВК 2565106. PMID 18941625.
- Сенапати, П. (1995). «Интроны и происхождение генов, кодирующих белок». Наука. 268 (5215): 1366–7. Bibcode:1995Научный ... 268.1366S. Дои:10.1126 / science.7761858. PMID 7761858.
Рекомендации
- ^ а б c d е ж грамм час я j k л м п о п q Senapathy, P. (апрель 1986 г.). «Происхождение эукариотических интронов: гипотеза, основанная на статистике распределения кодонов в генах, и ее последствия». Труды Национальной академии наук Соединенных Штатов Америки. 83 (7): 2133–2137. Bibcode:1986ПНАС ... 83.2133С. Дои:10.1073 / pnas.83.7.2133. ISSN 0027-8424. ЧВК 323245. PMID 3457379.
- ^ а б c d е ж грамм час я j k л м п о Сенапати, П. (февраль 1982 г.). «Возможная эволюция сигналов сплайс-соединения в генах эукариот из стоп-кодонов». Труды Национальной академии наук Соединенных Штатов Америки. 85 (4): 1129–1133. Bibcode:1988ПНАС ... 85.1129С. Дои:10.1073 / pnas.85.4.1129. ISSN 0027-8424. ЧВК 279719. PMID 3422483.
- ^ а б c d е ж грамм час я j Сенапати, П. (1995-06-02). «Интроны и происхождение генов, кодирующих белок». Наука. 268 (5215): 1366–1367, ответ автора 1367–1369. Bibcode:1995Научный ... 268.1366S. Дои:10.1126 / science.7761858. ISSN 0036-8075. PMID 7761858.
- ^ Теобальд, Дуглас Л. (2012). «Более 29 свидетельств макроэволюции: научное обоснование общего происхождения». Цитировать журнал требует
| журнал =(помощь) - ^ Gillies, S.D .; Morrison, S.L .; Ои, В. Т .; Тонегава, С. (июнь 1983 г.). «Тканеспецифический элемент энхансера транскрипции расположен в главном интроне реаранжированного гена тяжелой цепи иммуноглобулина». Клетка. 33 (3): 717–728. Дои:10.1016/0092-8674(83)90014-4. ISSN 0092-8674. PMID 6409417.
- ^ Mercola, M .; Wang, X. F .; Olsen, J .; Каламе, К. (1983-08-12). «Элементы усилителя транскрипции в локусе тяжелой цепи иммуноглобулина мыши». Наука. 221 (4611): 663–665. Bibcode:1983Наука ... 221..663М. Дои:10.1126 / science.6306772. ISSN 0036-8075. PMID 6306772.
- ^ Берк, А. Дж .; Шарп П. А. (ноябрь 1977 г.). «Определение размеров и картирование мРНК ранних аденовирусов с помощью гель-электрофореза гибридов, расщепленных эндонуклеазой S1». Клетка. 12 (3): 721–732. Дои:10.1016/0092-8674(77)90272-0. ISSN 0092-8674. PMID 922889.
- ^ Бергет, С. М.; Мур, C; Шарп, Пенсильвания (август 1977 г.). «Сплайсированные сегменты на 5'-конце поздней мРНК аденовируса 2». Труды Национальной академии наук Соединенных Штатов Америки. 74 (8): 3171–3175. Bibcode:1977ПНАС ... 74.3171Б. Дои:10.1073 / pnas.74.8.3171. ISSN 0027-8424. ЧВК 431482. PMID 269380.
- ^ Чоу, Л. Т .; Робертс, Дж. М .; Lewis, J. B .; Брокер Т. Р. (август 1977 г.). «Карта цитоплазматических транскриптов РНК литического аденовируса типа 2, определенная с помощью электронной микроскопии гибридов РНК: ДНК». Клетка. 11 (4): 819–836. Дои:10.1016 / 0092-8674 (77) 90294-Х. ISSN 0092-8674. PMID 890740.
- ^ «Комплект для онлайн-обучения: 1977: обнаружены интроны». Национальный институт исследования генома человека (NHGRI). Получено 2019-01-01.
- ^ Дулиттл, У. Форд (13 апреля 1978 г.). «Гены по частям: были ли они когда-нибудь вместе?». Природа. 272 (5654): 581–582. Bibcode:1978Натура.272..581D. Дои:10.1038 / 272581a0. ISSN 1476-4687.
- ^ Дарнелл, Дж. Э. (1978-12-22). «Влияние сплайсинга РНК-РНК в эволюции эукариотических клеток». Наука. 202 (4374): 1257–1260. Дои:10.1126 / science.364651. ISSN 0036-8075. PMID 364651.
- ^ Дулиттл, В. Ф .; Дарнелл, Дж. Э. (1986-03-01). «Размышления о раннем ходе эволюции». Труды Национальной академии наук. 83 (5): 1271–1275. Bibcode:1986ПНАС ... 83.1271Д. Дои:10.1073 / pnas.83.5.1271. ISSN 1091-6490. ЧВК 323057. PMID 2419905.
- ^ а б c Блейк, C.C.F. (1985-01-01). Экзоны и эволюция белков. Международный обзор цитологии. 93. С. 149–185. Дои:10.1016 / S0074-7696 (08) 61374-1. ISBN 9780123644930. ISSN 0074-7696.
- ^ Гилберт, Уолтер (февраль 1978 г.). «Почему гены по частям?». Природа. 271 (5645): 501. Bibcode:1978Натура.271..501Г. Дои:10.1038 / 271501a0. ISSN 1476-4687. PMID 622185.
- ^ Тонегава, S; Максам, А М; Тизард, Р; Бернар, О; Гилберт, Вт (март 1978 г.). «Последовательность гена зародышевой линии мыши для вариабельной области легкой цепи иммуноглобулина». Труды Национальной академии наук Соединенных Штатов Америки. 75 (3): 1485–1489. Bibcode:1978PNAS ... 75.1485T. Дои:10.1073 / пнас.75.3.1485. ISSN 0027-8424. ЧВК 411497. PMID 418414.
- ^ Feng, D. F .; Дулиттл, Р. Ф. (1 января 1987 г.). «Реконструкция эволюции свертывания крови позвоночных с учетом аминокислотных последовательностей белков свертывания». Симпозиумы Колд-Спринг-Харбор по количественной биологии. 52: 869–874. Дои:10.1101 / SQB.1987.052.01.095. ISSN 1943-4456. PMID 3483343.
- ^ Гиббонс, А. (1990-12-07). «Расчет исходного семейства - экзонов». Наука. 250 (4986): 1342. Bibcode:1990Sci ... 250.1342G. Дои:10.1126 / science.1701567. ISSN 1095-9203. PMID 1701567.
- ^ а б c Рева, Олег; Тюммлер, Буркхард (2008). «Думайте масштабно - гигантские гены у бактерий» (PDF). Экологическая микробиология. 10 (3): 768–777. Дои:10.1111 / j.1462-2920.2007.01500.x. HDL:2263/9009. ISSN 1462-2920. PMID 18237309.
- ^ а б c d е ж грамм Регулапати, Рахул; Сингх, Чандан Кумар; Бхаси, Ашвини; Senapathy, Periannan (2008-10-20). «Происхождение расщепленной структуры сплайсосомных генов из случайных генетических последовательностей». PLOS ONE. 3 (10): e3456. Bibcode:2008PLoSO ... 3.3456R. Дои:10.1371 / journal.pone.0003456. ISSN 1932-6203. ЧВК 2565106. PMID 18941625.
- ^ Информация, Reed Business (1986-06-26). Новый ученый. Деловая информация компании Reed.
- ^ Информация, Reed Business (1988-03-31). Новый ученый. Деловая информация компании Reed.
- ^ а б c d Сенапати, перианнан; Харрис, Номи Л. (1990-05-25). «Распределение и согласование сигналов точек ветвления в эукариотических генах: компьютеризированный статистический анализ». Исследования нуклеиновых кислот. 18 (10): 3015–9. Дои:10.1093 / nar / 18.10.3015. ISSN 0305-1048. ЧВК 330832. PMID 2349097.
- ^ Maier, U.-G .; Браун, J.W.S .; Toloczyki, C .; Фикс, Г. (январь 1987 г.). «Связывание ядерного фактора с консенсусной последовательностью в 5'-фланкирующей области генов зеина кукурузы». Журнал EMBO. 6 (1): 17–22. Дои:10.1002 / j.1460-2075.1987.tb04712.x. ISSN 0261-4189. ЧВК 553350. PMID 15981330.
- ^ Keller, E B; Полдень, Вашингтон (1985-07-11). «Сплайсинг интронов: консервативный внутренний сигнал в интронах пре-мРНК дрозофилы». Исследования нуклеиновых кислот. 13 (13): 4971–4981. Дои:10.1093 / nar / 13.13.4971. ISSN 0305-1048. ЧВК 321838. PMID 2410858.
- ^ БИРНСТИЛЬ, М; БУСЛИНГЕР, М; СТРУБ, К. (июнь 1985 г.). «Прерывание транскрипции и 3 'обработка: конец на месте!». Клетка. 41 (2): 349–359. Дои:10.1016 / с0092-8674 (85) 80007-6. ISSN 0092-8674.
- ^ Консорциум, Международное секвенирование генома человека (февраль 2001 г.). "Начальная последовательность и анализ человеческого генома". Природа. 409 (6822): 860–921. Bibcode:2001Натура.409..860л. Дои:10.1038/35057062. ISSN 1476-4687. PMID 11237011.
- ^ Чжу, Сяохун; Зандие, Али; Ся, Эшли; Ву, Митчелл; Ву, Дэвид; Вэнь, Мэйюань; Ван, Мэй; Вентер, Эли; Тернер, Рассел (2001-02-16). «Последовательность генома человека». Наука. 291 (5507): 1304–1351. Bibcode:2001Научный ... 291.1304V. Дои:10.1126 / science.1058040. ISSN 1095-9203. PMID 11181995.
- ^ Канг, Бёнг-Чорл; Нет, Гёнджу; Ли, Хын-Рюль; Хан, Койн; Purushotham, Preethi M .; Джо, Джинкван (2017). «Разработка генетической карты лука (Allium cepa L.) с использованием безреференсного генотипирования путем секвенирования и анализов SNP». Границы растениеводства. 8: 1606. Дои:10.3389 / fpls.2017.01606. ISSN 1664-462X. ЧВК 5604068. PMID 28959273.
- ^ Smith, Jeramiah J .; Восс, С. Рэндал; Tsonis, Panagiotis A .; Тимошевская Наталия Юрьевна; Тимошевский, Владимир А .; Кейнат, Мелисса К. (10 ноября 2015 г.). «Первоначальная характеристика большого генома саламандры Ambystoma mexicanum с использованием дробовика и секвенирования хромосом с лазерным захватом». Научные отчеты. 5: 16413. Bibcode:2015НатСР ... 516413K. Дои:10.1038 / srep16413. ISSN 2045-2322. ЧВК 4639759. PMID 26553646.
- ^ Venter, J.C .; Adams, M.D .; Myers, E.W .; Li, P.W .; Mural, R.J .; Sutton, G.G .; Smith, H.O .; Yandell, M .; Эванс, К. А. (16 февраля 2001 г.). «Последовательность генома человека». Наука. 291 (5507): 1304–1351. Bibcode:2001Научный ... 291.1304V. Дои:10.1126 / science.1058040. ISSN 0036-8075. PMID 11181995.
- ^ Lander, E. S .; Linton, L.M .; Birren, B .; Nusbaum, C .; Zody, M. C .; Болдуин, Дж .; Девон, К .; Dewar, K .; Дойл, М. (15 февраля 2001 г.). "Начальная последовательность и анализ человеческого генома" (PDF). Природа. 409 (6822): 860–921. Bibcode:2001Натура.409..860л. Дои:10.1038/35057062. ISSN 0028-0836. PMID 11237011.
- ^ Консорциум *, Секвенирование C. elegans (1998-12-11). «Последовательность генома нематоды C. elegans: платформа для изучения биологии». Наука. 282 (5396): 2012–2018. Bibcode:1998На ... 282.2012.. Дои:10.1126 / science.282.5396.2012. ISSN 1095-9203. PMID 9851916.
- ^ Инициатива по геному арабидопсиса (2000-12-14). «Анализ последовательности генома цветкового растения Arabidopsis thaliana». Природа. 408 (6814): 796–815. Bibcode:2000Натура 408..796Т. Дои:10.1038/35048692. ISSN 0028-0836. PMID 11130711.
- ^ Беннетцен, Джеффри Л .; Браун, Джеймс К. М .; Девос, Катриен М. (01.07.2002). «Уменьшение размера генома за счет незаконной рекомбинации противодействует расширению генома у Arabidopsis». Геномные исследования. 12 (7): 1075–1079. Дои:10.1101 / гр.132102. ISSN 1549-5469. ЧВК 186626. PMID 12097344.
- ^ Курляндия, C.G .; Canbäck, B .; Берг, О. Г. (декабрь 2007 г.). «Истоки современных протеомов». Биохимия. 89 (12): 1454–1463. Дои:10.1016 / j.biochi.2007.09.004. ISSN 0300-9084. PMID 17949885.
- ^ Каэтано-Аноллес, Густаво; Каэтано-Аноллес, Дерек (июль 2003 г.). «Эволюционно структурированная вселенная белковой архитектуры». Геномные исследования. 13 (7): 1563–1571. Дои:10.1101 / гр.1161903. ISSN 1088-9051. ЧВК 403752. PMID 12840035.
- ^ Глансдорф, Николас; Сюй, Инь; Лабедан, Бернард (2008-07-09). «Последний универсальный общий предок: возникновение, конституция и генетическое наследие неуловимого предшественника». Биология Директ. 3: 29. Дои:10.1186/1745-6150-3-29. ISSN 1745-6150. ЧВК 2478661. PMID 18613974.
- ^ Курляндия, C.G .; Коллинз, Л. Дж .; Пенни, Д. (19 мая 2006 г.). «Геномика и несводимая природа эукариотических клеток». Наука. 312 (5776): 1011–1014. Bibcode:2006Научный ... 312.1011K. Дои:10.1126 / science.1121674. ISSN 1095-9203. PMID 16709776.
- ^ Коллинз, Лесли; Пенни, Дэвид (апрель 2005 г.). «Сложная сплайсосомная организация предков современных эукариот». Молекулярная биология и эволюция. 22 (4): 1053–1066. Дои:10.1093 / molbev / msi091. ISSN 0737-4038. PMID 15659557.
- ^ Пенни, Дэвид; Коллинз, Лесли Дж .; Дейли, Тони К .; Кокс, Саймон Дж. (Декабрь 2014 г.). «Относительный возраст эукариот и акариотов». Журнал молекулярной эволюции. 79 (5–6): 228–239. Bibcode:2014JMolE..79..228P. Дои:10.1007 / s00239-014-9643-у. ISSN 1432-1432. PMID 25179144.
- ^ Fuerst, John A .; Сагуленко, Евгений (04.05.2012). «Ключи к эукариальности: планктомицеты и наследственная эволюция клеточной сложности». Границы микробиологии. 3: 167. Дои:10.3389 / fmicb.2012.00167. ISSN 1664-302X. ЧВК 3343278. PMID 22586422.
- ^ Коллинз, Лесли; Пенни, Дэвид (апрель 2005 г.). «Сложная сплайсосомная организация предков современных эукариот». Молекулярная биология и эволюция. 22 (4): 1053–1066. Дои:10.1093 / molbev / msi091. ISSN 0737-4038. PMID 15659557.[требуется проверка ]
- ^ а б c d Шапиро, М.Б .; Сенапати, П. (1987-09-11). «Соединения сплайсинга РНК различных классов эукариот: статистика последовательностей и функциональное значение в экспрессии генов». Исследования нуклеиновых кислот. 15 (17): 7155–7174. Дои:10.1093 / nar / 15.17.7155. ISSN 0305-1048. ЧВК 306199. PMID 3658675.
- ^ а б c d Senapathy, P .; Шапиро, М.Б .; Харрис, Н. Л. (1990). Соединения сплайсинга, сайты точек ветвления и экзоны: статистика последовательностей, идентификация и приложения в геномном проекте. Методы в энзимологии. 183. С. 252–278. Дои:10.1016/0076-6879(90)83018-5. ISBN 9780121820848. ISSN 0076-6879. PMID 2314278.
- ^ «Национальные институты здоровья (NIH) - все мы». allofus.nih.gov. Получено 2019-01-02.
- ^ а б Пенни, Дэвид; Коллинз, Лесли (1 апреля 2005 г.). «Сложная сплайсосомная организация, являющаяся предком существующих эукариот». Молекулярная биология и эволюция. 22 (4): 1053–1066. Дои:10.1093 / molbev / msi091. ISSN 0737-4038. PMID 15659557.
- ^ Каэтано-Аноллес, Дерек; Каэтано-Аноллес, Густаво (1 июля 2003 г.). «Эволюционно структурированная вселенная белковой архитектуры». Геномные исследования. 13 (7): 1563–1571. Дои:10.1101 / гр.1161903. ISSN 1549-5469. ЧВК 403752. PMID 12840035.
- ^ Глансдорф, Николас; Сюй, Инь; Лабедан, Бернард (2008-07-09). «Последний всеобщий общий предок: возникновение, конституция и генетическое наследие неуловимого предшественника». Биология Директ. 3 (1): 29. Дои:10.1186/1745-6150-3-29. ISSN 1745-6150. ЧВК 2478661. PMID 18613974.
- ^ Курляндия, C.G .; Canbäck, B .; Берг, О. (2007-12-01). «Истоки современных протеомов». Биохимия. 89 (12): 1454–1463. Дои:10.1016 / j.biochi.2007.09.004. ISSN 0300-9084. PMID 17949885.
- ^ Penny, D .; Коллинз, Л. Дж .; Курланд, К. Г. (19 мая 2006 г.). «Геномика и неприводимая природа клеток эукариот». Наука. 312 (5776): 1011–1014. Bibcode:2006Научный ... 312.1011K. Дои:10.1126 / science.1121674. ISSN 1095-9203. PMID 16709776.
- ^ Poole, A.M .; Джеффарес, Д. С .; Пенни, Д. (январь 1998 г.). «Путь из мира РНК». Журнал молекулярной эволюции. 46 (1): 1–17. Bibcode:1998JMolE..46 .... 1P. Дои:10.1007 / PL00006275. ISSN 0022-2844. PMID 9419221.
- ^ Фортер, Патрик; Филипп, Эрве (1999). «Где корень вселенского древа жизни?». BioEssays. 21 (10): 871–879. Дои:10.1002 / (SICI) 1521-1878 (199910) 21:10 <871 :: AID-BIES10> 3.0.CO; 2-Q. ISSN 1521-1878. PMID 10497338.
- ^ Кокс, Саймон Дж .; Дали, Тони К .; Коллинз, Лесли Дж .; Пенни, Дэвид (2014-12-01). «Относительный возраст эукариот и акариот». Журнал молекулярной эволюции. 79 (5–6): 228–239. Bibcode:2014JMolE..79..228P. Дои:10.1007 / s00239-014-9643-у. ISSN 1432-1432. PMID 25179144.
- ^ Сагуленко, Евгений; Фуэрст, Джон Арлингтон (2012). «Ключи к эукариальности: планктомицеты и наследственная эволюция клеточной сложности». Границы микробиологии. 3. Дои:10.3389 / fmicb.2012.00167. ISSN 1664-302X. ЧВК 3343278. PMID 22586422.
- ^ а б Гилберт, Уолтер; Рой, Скотт В. (2005-02-08). «Сложные ранние гены». Труды Национальной академии наук. 102 (6): 1986–1991. Bibcode:2005ПНАС..102.1986Р. Дои:10.1073 / pnas.0408355101. ISSN 1091-6490. ЧВК 548548. PMID 15687506.
- ^ Гилберт, Уолтер; Рой, Скотт Уильям (март 2006 г.). «Эволюция сплайсосомных интронов: закономерности, загадки и прогресс». Природа Обзоры Генетика. 7 (3): 211–221. Дои:10.1038 / nrg1807. ISSN 1471-0064. PMID 16485020.
- ^ Рогозин, Игорь Б .; Свердлов, Александр В .; Бабенко, Владимир Н .; Кунин, Евгений В. (июнь 2005 г.). «Анализ эволюции экзон-интронной структуры эукариотических генов». Брифинги по биоинформатике. 6 (2): 118–134. Дои:10.1093 / bib / 6.2.118. ISSN 1467-5463. PMID 15975222.
- ^ Салливан, Джеймс С.; Reitzel, Adam M .; Финнерти, Джон Р. (2006). «Высокий процент интронов в генах человека присутствовал на ранних этапах эволюции животных: свидетельства от базального многоклеточного животного Nematostella vectensis». Геномная информатика. Международная конференция по геномной информатике. 17 (1): 219–229. ISSN 0919-9454. PMID 17503371.
- ^ Кунин, Евгений В .; Рогозин, Игорь Б .; Чурос, Миклош (15.09.2011). «Подробная история богатых интроном эукариотических предков, полученная в результате глобального исследования 100 полных геномов». PLOS вычислительная биология. 7 (9): e1002150. Bibcode:2011PLSCB ... 7E2150C. Дои:10.1371 / journal.pcbi.1002150. ISSN 1553-7358. ЧВК 3174169. PMID 21935348.
