Просвечивающая электронная микроскопия, секвенирование ДНК - Transmission electron microscopy DNA sequencing
Просвечивающая электронная микроскопия, секвенирование ДНК это простая молекула последовательность действий технология, которая использует просвечивающая электронная микроскопия техники. Метод был задуман и разработан в 1960-х и 1970-х годах.[1] но потерял благосклонность, когда был признан размер повреждения образца.[2]
Чтобы ДНК четко визуализировалась под электронный микроскоп, он должен быть помечен тяжелыми атомами. Кроме того, специальные методы визуализации и аберрация исправлена оптика полезна для получения разрешающая способность требуется для изображения меченой молекулы ДНК. Теоретически секвенирование ДНК с помощью просвечивающей электронной микроскопии может обеспечить чрезвычайно большую длину считывания, но проблема повреждения электронного луча все еще может оставаться, а технология еще не получила коммерческого развития.
История
Всего несколько лет спустя Джеймс Уотсон и Фрэнсис Крик вывел структура ДНК, и почти два десятилетия назад Фредерик Сэнгер опубликовал первый метод быстрого Секвенирование ДНК, Ричард Фейнман, американский физик, предвидел электронный микроскоп как инструмент, который однажды позволит биологам «увидеть порядок базы в ДНК цепь".[3] Фейнман считал, что если электронный микроскоп можно сделать достаточно мощным, то станет возможным визуализировать атомную структуру любых химических соединений, включая ДНК.
В 1970 г. Альберт Крю разработал большой угол кольцевое изображение в темном поле (HAADF) метод визуализации в растровый просвечивающий электронный микроскоп. Используя эту технику, он визуализировал отдельные тяжелые атомы на тонких пленках аморфного углерода.[4] В 2010 году Криванек и его коллеги сообщили о нескольких технических усовершенствованиях метода HAADF, включая сочетание электронной оптики с коррекцией аберраций и низкого ускоряющего напряжения. Последнее имеет решающее значение для визуализации биологических объектов, так как позволяет уменьшить повреждение луча и увеличить контраст изображения для легких атомов. В результате можно было отобразить замещения отдельных атомов в монослое нитрида бора.[5]
Несмотря на изобретение множества технологий химического и флуоресцентного секвенирования, электронная микроскопия все еще исследуется как средство выполнения секвенирования одномолекулярной ДНК. Например, в 2012 году сотрудничество ученых из Гарвардский университет, то Университет Нью-Гэмпшира и ZS Genetics продемонстрировал способность читать длинные последовательности ДНК с помощью этой техники,[6] однако технология секвенирования ДНК с помощью просвечивающей электронной микроскопии все еще далека от коммерческой доступности.[7]
Принцип
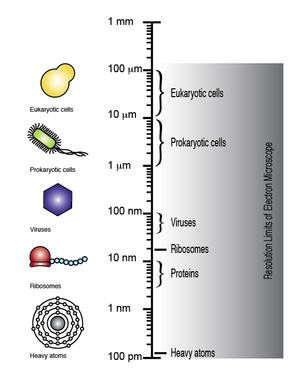
В электронный микроскоп обладает способностью получать разрешение до 100 мкм, благодаря чему можно наблюдать микроскопические биомолекулы и структуры, такие как вирусы, рибосомы, белки, липиды, небольшие молекулы и даже отдельные атомы.[8]
Несмотря на то что ДНК видно при наблюдении в электронный микроскоп, разрешение полученного изображения недостаточно высоко, чтобы можно было расшифровать последовательность отдельных базы, т.е., Секвенирование ДНК. Однако при дифференциальной маркировке оснований ДНК тяжелыми атомами или металлами можно как визуализировать, так и различать отдельные основания. Следовательно, электронная микроскопия в сочетании с дифференциальной маркировкой тяжелого атома ДНК можно использовать для прямого изображения ДНК с целью определения ее последовательности.[7][9][10][11]
Рабочий процесс
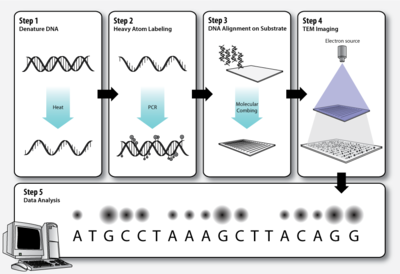
Шаг 1 - денатурация ДНК
Как в стандарте полимеразная цепная реакция (ПЦР), секвенируемые двухцепочечные молекулы ДНК должны быть денатурированный до того, как вторая цепь может быть синтезирована с мечеными нуклеотидами.
Шаг 2 - Мечение тяжелых атомов
Элементы, из которых состоят биологические молекулы (C, ЧАС, N, О, п, S ) слишком легкие (низкие атомный номер, Z ), чтобы они были четко визуализированы как отдельные атомы просвечивающая электронная микроскопия. Чтобы обойти эту проблему, ДНК базы может быть помечен более тяжелыми атомами (более высокое Z). Каждый нуклеотид помечены характерной жирной меткой, чтобы их можно было различить на микрофотографии просвечивающего электронного микроскопа.
- ZS Genetics предлагает использовать три тяжелых лейбла: бром (Z = 35), йод (Z = 53) и трихлорметан (всего Z = 63). На микрофотографии они будут выглядеть как разные темные и светлые пятна, а четвертое основание ДНК останется немаркированным.
- Halcyon Molecular в сотрудничестве с группой Toste предлагает, чтобы пурин и пиримидин основания могут быть функционализированы диамином платины или бипиридином тетраоксида осмия соответственно. Атомы тяжелых металлов, такие как осмий (Z = 76), иридий (Z = 77), золото (Z = 79), или уран (Z = 92) может затем образовывать связи металл-металл с этими функциональными группами для метки индивидуальных оснований.[12]
Шаг 3 - выравнивание ДНК на субстрате
Молекулы ДНК необходимо растянуть на тонкой твердой подложке, чтобы порядок помеченных оснований был четко виден на электронной микрофотографии. Молекулярное расчесывание это метод, который использует силу удаляющейся границы раздела воздух-вода для удлинения молекул ДНК, оставляя их необратимо связанными со слоем силана после высыхания.[13][14] Это одно из средств, с помощью которого может быть достигнуто выравнивание ДНК на твердом субстрате.
Шаг 4 - визуализация ПЭМ

Просвечивающая электронная микроскопия (ПЭМ) дает большое увеличение, изображения с высоким разрешением пропуская пучок электронов через очень тонкий образец. В то время как атомное разрешение было продемонстрировано с помощью обычного ПЭМ, дальнейшее улучшение пространственного разрешения требует корректировки сферический и хроматические аберрации микроскопа линзы. Это было возможно только в растровая просвечивающая электронная микроскопия где изображение получается путем сканирования объекта тонко сфокусированным электронным лучом аналогично электронно-лучевая трубка. Однако достигнутое улучшение разрешения сопровождается облучением исследуемого объекта гораздо более высокой интенсивностью луча, сопутствующим повреждением образца и связанными с ним артефактами изображения.[15] В зависимости от того, содержит ли образец тяжелые или легкие атомы, применяются различные методы визуализации:
- Кольцевая визуализация темного поля измеряет рассеяние электронов, когда они отклоняются от ядер атомов в образце просвечивающей электронной микроскопии.[5] Это лучше всего подходит для образцов, содержащих тяжелые атомы, поскольку они вызывают большее рассеяние электронов. Этот метод использовался для изображения атомов такими светлыми, как бор, азот, и углерод;[5] однако для таких легких атомов сигнал очень слабый. Если кольцевую темнопольную микроскопию использовать для секвенирования ДНК с помощью просвечивающей электронной микроскопии, безусловно, необходимо будет пометить основания ДНК тяжелыми атомами, чтобы можно было обнаружить сильный сигнал.
- Кольцевая визуализация в светлом поле обнаруживает электроны, проходящие непосредственно через образец, и измеряет интерференцию волн, создаваемую их взаимодействием с атомными ядрами. Этот метод может обнаруживать легкие атомы с большей чувствительностью, чем методы кольцевой визуализации темного поля. Фактически, кислород,[16] азот,[16] литий,[17] и водород[18] в кристаллических твердых телах были получены с помощью кольцевой электронной микроскопии в светлом поле. Таким образом, теоретически возможно получить прямые изображения атомов в цепи ДНК; однако структура ДНК намного менее геометрическая, чем у кристаллических твердых тел, поэтому прямая визуализация без предварительного мечения может быть невозможна.
Шаг 5 - Анализ данных
Темные и светлые пятна на электронной микрофотографии, соответствующие различным основам ДНК, анализируются компьютерным программным обеспечением.
Приложения
Секвенирование ДНК с помощью просвечивающей электронной микроскопии еще не коммерчески доступно, но большая длина считывания, которую эта технология может однажды обеспечить, сделает ее полезной в различных контекстах.
De novo сборка генома
При секвенировании генома его необходимо разбить на части, достаточно короткие, чтобы их можно было секвенировать за одно считывание. Затем эти чтения необходимо собрать вместе, как мозаику, выровняв области, перекрывающиеся между чтениями; этот процесс называется de novo сборка генома. Чем больше длина считывания, которую предоставляет платформа для секвенирования, тем длиннее перекрывающиеся области и тем легче собрать геном. С вычислительной точки зрения, микрофлюидное секвенирование по Сэнгеру по-прежнему является наиболее эффективным способом секвенирования и сборки геномов, для которых нет эталонный геном последовательность существует. Относительно большие длины считывания обеспечивают существенное перекрытие между отдельными считываниями секвенирования, что позволяет повысить статистическую достоверность сборки. Кроме того, длинные чтения по Сэнгеру могут охватывать большинство регионов повторяющаяся последовательность ДНК которые в противном случае затрудняют сборку последовательности, вызывая ложное выравнивание. Тем не мение, de novo Сборка генома с помощью секвенирования по Сэнгеру чрезвычайно дорога и требует много времени. Технологии секвенирования второго поколения,[19] в то время как менее дорогие, обычно непригодны для de novo сборка генома из-за коротких длин чтения. В целом технологии секвенирования третьего поколения,[11] включая секвенирование ДНК с помощью просвечивающей электронной микроскопии, чтобы увеличить длину считывания при сохранении низкой стоимости секвенирования. Таким образом, по мере совершенствования технологий секвенирования третьего поколения, быстрое и недорогое de novo сборка генома станет реальностью.
Полные гаплотипы
А гаплотип это серия связанных аллели которые наследуются вместе на одной хромосоме. Секвенирование ДНК можно использовать для генотип Все однонуклеотидные полиморфизмы (SNP) которые составляют гаплотип. Однако короткие чтения секвенирования ДНК часто не могут быть поэтапными; то есть, гетерозиготный варианты не могут быть с уверенностью отнесены к правильному гаплотипу. Фактически, гаплотипирование с помощью данных секвенирования ДНК с коротким считыванием требует очень высокого охвата (в среднем> 50-кратного охвата каждой базы ДНК) для точной идентификации SNP, а также дополнительных данных последовательности от родителей, чтобы Менделирующая передача можно использовать для оценки гаплотипов.[20] Технологии секвенирования, которые генерируют длинные считывания, включая секвенирование ДНК с помощью просвечивающей электронной микроскопии, могут захватывать целые гаплоблоки за одно считывание. То есть гаплотипы не разбиваются между множественными считываниями, и генетически связанные аллели остаются вместе в данных секвенирования. Таким образом, длинные чтения делают гаплотипирование более простым и точным, что полезно для популяционная генетика.
Копировать варианты номеров
Гены обычно присутствуют в двух копиях в диплоид человеческий геном; гены, которые отклоняются от этого стандартного числа копий, называются варианты числа копий (CNV). Вариация числа копий может быть доброкачественной (обычно это распространенные варианты, называемые полиморфизмом числа копий) или патогенными.[21] CNV обнаруживаются флуоресцентная гибридизация in situ (FISH) или же сравнительная геномная гибридизация (CGH). Для обнаружения конкретных контрольных точек, в которых происходит делеция, или для обнаружения геномных повреждений, вызванных событием дупликации или амплификации, CGH может быть выполнен с использованием мозаичный массив (массив CGH ), или вариантная область может быть секвенирована. Длинные считывания секвенирования особенно полезны для анализа дупликаций или амплификаций, поскольку можно проанализировать ориентацию амплифицированных сегментов, если они были захвачены в одном считывании секвенирования.
Рак
Геномика рака, или онкогеномика, это новая область, в которой высокопроизводительное секвенирование ДНК второго поколения технология применяется для секвенирования целых геномов рака. Анализ этих данных последовательности коротких чтений охватывает все проблемы, связанные с de novo сборка генома с использованием коротких данных чтения.[22] Кроме того, геномы рака часто анеуплоидный.[23] Эти аберрации, которые по существу являются крупномасштабными вариантами числа копий, могут быть проанализированы с помощью технологий секвенирования второго поколения, используя частоту считывания для оценки числа копий.[22] Однако более длинные считывания дадут более точную картину количества копий, ориентации амплифицированных областей и SNP, присутствующих в геномах рака.
Секвенирование микробиома
В микробиом относится к общей коллекции микробов, присутствующих в микросреде, и их соответствующих геномах. Например, около 100 триллионов микробных клеток колонизируют человеческое тело в любой момент времени.[24] Микробиом человека представляет особый интерес, так как эти комменсальные бактерии важны для здоровья и иммунитета человека. Большинство бактериальных геномов Земли еще не секвенировано; осуществление проекта по секвенированию микробиома потребует обширных de novo сборка генома, перспектива, которая устрашает с технологиями короткого считывания последовательности ДНК.[25] Более длинные чтения значительно упростят сборку новых микробных геномов.
Сильные и слабые стороны
По сравнению с другими технологиями секвенирования ДНК второго и третьего поколений секвенирование ДНК с помощью просвечивающей электронной микроскопии имеет ряд потенциальных ключевых сильных и слабых сторон, которые в конечном итоге определят его полезность и известность как будущую технологию секвенирования ДНК.
Сильные стороны
- Более длинные чтения: ZS Genetics оценила потенциальную длину считывания секвенирования ДНК с помощью просвечивающей электронной микроскопии от 10 000 до 20 000 пар оснований со скоростью 1,7 миллиарда пар оснований в день.[7] Такая большая длина чтения позволит легче de novo сборка генома и прямое обнаружение гаплотипов, среди прочего.[11]
- Низкая стоимость: Трансмиссионная электронная микроскопия. По оценкам, секвенирование ДНК обходится всего в 5-10 000 долларов США на геном человека по сравнению с более дорогими альтернативами секвенирования ДНК второго поколения.[10]
- Без дефазировки: Дефазирование цепей ДНК из-за потери синхронности во время синтеза является основной проблемой технологий секвенирования второго поколения. Для секвенирования ДНК с помощью просвечивающей электронной микроскопии и нескольких других технологий секвенирования третьего поколения синхронизация считываний не требуется, поскольку одновременно считывается только одна молекула.[7][11]
- Более короткие сроки выполнения работ: Возможность считывать нативные фрагменты ДНК делает подготовку сложной матрицы ненужным шагом в общем рабочем процессе секвенирования всего генома. Следовательно, возможно более короткое время выполнения работ.[11]
Недостатки
- Высокая капитальная стоимость: Просвечивающий электронный микроскоп с достаточным разрешением, необходимым для просвечивающей электронной микроскопии. Секвенирование ДНК стоит около 1000000 долларов США, поэтому проведение секвенирования ДНК этим методом требует значительных инвестиций.[10]
- Технически сложно: Селективное маркирование тяжелых атомов, прикрепление и выпрямление меченой ДНК к субстрату является серьезной технической проблемой.[10] Кроме того, образец ДНК должен быть устойчивым к высокому вакууму электронного микроскопа и облучению сфокусированным пучком электронов высокой энергии.
- Потенциал ПЦР предвзятость и артефакты: Хотя ПЦР используется только при секвенировании ДНК с помощью просвечивающей электронной микроскопии как средство маркировки цепи ДНК тяжелыми атомами или металлами, может существовать возможность внесения смещения в представление матрицы или ошибок во время однократной амплификации.[10]
Сравнение с другими технологиями секвенирования
Многие технологии секвенирования ДНК второго и третьего поколений, не относящиеся к Сэнгеру, были разработаны или в настоящее время разрабатываются с общей целью увеличения пропускной способности и снижения стоимости, чтобы можно было полностью реализовать персонализированную генетическую медицину.
И 10 миллионов долларов США Приз Archon X для геномики при поддержке X Prize Foundation (Санта-Моника, Калифорния, США) и гранты в размере 70 миллионов долларов США при поддержке Национальный институт исследования генома человека из Национальные институты здоровья (NIH-NHGRI) способствуют быстрому всплеску исследовательской деятельности по разработке новых технологий секвенирования ДНК.[7]
Поскольку разные подходы, методы и стратегии определяют каждую технологию секвенирования ДНК, у каждой есть свои сильные и слабые стороны. Сравнение важных параметров между различными технологиями секвенирования ДНК второго и третьего поколений представлено в таблице 1.
| Платформа | Поколение | Длина чтения (бит) | Точность | Стоимость генома человека (долл. США) | Стоимость инструмента (долл. США) | Время работы (ч / Гбит)[7] |
|---|---|---|---|---|---|---|
| Массивно-параллельное пиросеквенирование путем синтеза | Второй | 400–500 | Q20 длина чтения 40 баз (99% при 400 базах и выше для предыдущих баз) | 1,000,000 | 500,000 | 75 |
| Секвенирование путем синтеза | Второй | 2×75 | Базовый вызов с Q30 (> 70%) | 60,000 | 450,000 | 56 |
| Секвенирование на основе массового параллельного клонального лигирования на основе гранул | Второй | 100 | 99.94% | 60,000 | 591,000 | 42 |
| Массивно-параллельное секвенирование одной молекулы путем синтеза | В третьих | 30–35 | 99,995% при> 20-кратном охвате (необработанная частота ошибок: ≤ 5%) | 70,000 | 1,350,000 | ~12 |
| Одиночная молекула, секвенирование в реальном времени путем синтеза | В третьих | 1000–1500 | 99,3% при 15-кратном охвате (частота ошибок при однократном считывании: 15–20%) | – | – | <1 |
| Секвенирование нанопор | В третьих | Потенциально без ограничений? | -- | -- | -- | >20 |
| Секвенирование отдельных молекул с помощью просвечивающей электронной микроскопии (ZS Genetics, Halcyon Molecular) | В третьих | Потенциально без ограничений? | -- | ~10,000 | ~1,000,000 | ~14 |
Рекомендации
- ^ [Майкл Бир и Ричард Зобель (1961) "Электронные пятна II: Электронно-микроскопические исследования видимости окрашенных молекул ДНК" J. Mol. Биол. Том 3, выпуск 6, декабрь 1961 г., страницы 717–726, IN3 – IN5 "]
- ^ [М. Cole et al. (1977) "Молекулярная микроскопия меченых полинуклеотидов: стабильность атомов осмия" J. Mol. Биол. Volume 117, Issue 2, 5 декабря 1977, страницы 387–400]
- ^ Фейнман Р. (1959) Внизу много места. Лекция в Калтехе.
- ^ Крю, Альберт V; Wall, J .; Лэнгмор, Дж. (1970). «Видимость отдельного атома». Наука. 168 (3937): 1338–1340. Bibcode:1970Sci ... 168.1338C. Дои:10.1126 / science.168.3937.1338. PMID 17731040. S2CID 31952480.
- ^ а б c Криванек О.Л .; Чисхолм, Мэтью Ф .; Николози, Валерия; Pennycook, Тимоти Дж .; Корбин, Джордж Дж .; Деллби, Никлас; Мерфитт, Мэтью Ф .; Собственный, Кристофер С .; Szilagyi, Zoltan S .; Оксли, Марк П .; Pantelides, Sokrates T .; Pennycook, Стивен Дж .; и другие. (2010). «Поатомный структурный и химический анализ методом кольцевой темнопольной электронной микроскопии». Природа. 464 (7288): 571–4. Bibcode:2010Натура.464..571K. Дои:10.1038 / природа08879. PMID 20336141. S2CID 1331554.
- ^ Белл Д., Томас В., Муртаг К., Дионн С., Грэм А., Андерсон Дж., Гловер В. (2012). «Идентификация оснований ДНК с помощью электронной микроскопии». Микроскопия и микроанализ. 18 (5): 1049–1053. Bibcode:2012MiMic..18.1049B. Дои:10.1017 / S1431927612012615. PMID 23046798.
- ^ а б c d е ж Гупта П.К. (2008). «Технологии секвенирования одномолекулярной ДНК для будущих исследований в области геномики». Тенденции в биотехнологии. 26 (11): 602–11. Дои:10.1016 / j.tibtech.2008.07.003. PMID 18722683.
- ^ Кэмпбелл Н.А. и Рис Дж. Б. (2002) Биология (6-е изд.). Сан-Франциско: Бенджамин Каммингс. ISBN 0-8053-6624-5
- ^ Уважаемый PH (2003). «Один за другим: инструменты одной молекулы для геномики». Брифинги по функциональной геномике и протеомике. 1 (4): 397–416. Дои:10.1093 / bfgp / 1.4.397. PMID 15239886.
- ^ а б c d е ж Сюй, М; Фудзита, Дайсуке; Ханагата, Нобутака; и другие. (2009). «Перспективы и проблемы новых технологий секвенирования одномолекулярной ДНК». Маленький. 5 (23): 2638–49. Дои:10.1002 / smll.200900976. PMID 19904762.
- ^ а б c d е Schadt EE; Тернер, С .; Касарскис, А .; и другие. (2010). «Окно в секвенирование третьего поколения». Молекулярная генетика человека. 19 (R2): R227-40. Дои:10,1093 / hmg / ddq416. PMID 20858600.
- ^ Награды за передовые технологии секвенирования 2010 г.. Genome.gov. Проверено 25 февраля 2011.
- ^ Бенсимон А; Саймон, А; Chiffaudel, A; Крокетт, V; Heslot, F; Bensimon, D; и другие. (1994). «Выравнивание и чувствительное обнаружение ДНК с помощью движущегося интерфейса». Наука. 265 (5181): 2096–8. Bibcode:1994Наука ... 265.2096Б. Дои:10.1126 / science.7522347. PMID 7522347.
- ^ Michalet X и др. (1997). «Динамическое молекулярное расчесывание: растяжение всего генома человека для исследований с высоким разрешением». Наука. 277 (5331): 1518–23. Дои:10.1126 / science.277.5331.1518. PMID 9278517. S2CID 22699914.
- ^ Haider M; Улеманн, Стефан; Шван, Ойген; Роза, Харальд; Кабиус, Бернд; Урбан, Кнут; и другие. (1998). "Электронно-микроскопическое изображение улучшено". Природа. 392 (6678): 768. Bibcode:1998Натура.392..768H. Дои:10.1038/33823. S2CID 205002987.
- ^ а б Okunishi E; Исикава, я; Савада, Н; Hosokawa, F; Хори, М; Кондо, Y; и другие. (2009). «Визуализация световых элементов в сверхвысоком разрешении с помощью кольцевой светопольной микроскопии STEM». Микроскопия и микроанализ. 15 (S2): 164–165. Bibcode:2009MiMic..15S.164O. Дои:10.1017 / S1431927609093891.
- ^ Осима Y; Sawada, H .; Hosokawa, F .; Okunishi, E .; Kaneyama, T .; Кондо, Й .; Niitaka, S .; Takagi, H .; Tanishiro, Y .; Такаянаги, К .; и другие. (2010). «Прямая визуализация атомов лития в LiV2О4 методом электронной микроскопии с коррекцией сферической аберрации ". Журнал электронной микроскопии. 59 (6): 457–61. Дои:10.1093 / jmicro / dfq017. PMID 20406731.
- ^ Ishikawa R; Окуниши, Эйдзи; Савада, Хидетака; Кондо, Юкихито; Хосокава, Фумио; Абэ, Эйдзи; и другие. (2011). «Прямая визуализация столбцов атомов водорода в кристалле с помощью кольцевой электронной микроскопии в светлом поле». Материалы Природы. 10 (4): 278–281. Bibcode:2011НатМа..10..278И. Дои:10.1038 / nmat2957. PMID 21317899.
- ^ Шендуре Дж, Джи Х (2008). «Секвенирование ДНК нового поколения». Природа Биотехнологии. 26 (10): 1135–45. Дои:10.1038 / nbt1486. PMID 18846087. S2CID 6384349.
- ^ Дурбин, Ричард М .; Альтшулер, Дэвид Л .; Дурбин, Ричард М .; Abecasis, Gonçalo R .; Бентли, Дэвид Р .; Чакраварти, Аравинда; Кларк, Эндрю Дж .; Коллинз, Фрэнсис С .; и другие. (2010). «Карта вариаций генома человека по результатам популяционного секвенирования». Природа. 467 (7319): 1061–73. Bibcode:2010 Натур.467.1061Т. Дои:10.1038 / природа09534. ЧВК 3042601. PMID 20981092.
- ^ Rodruiguez-Revenga L .; Мила, Монтсеррат; Розенберг, Карла; Лэмб, Аллен; Ли, Чарльз; и другие. (2007). «Структурные вариации в геноме человека: влияние вариантов числа копий на клинический диагноз». Генетика в медицине. 9 (9): 600–6. Дои:10.1097 / GIM.0b013e318149e1e3. PMID 17873648.
- ^ а б Такер Т; Марра, Марко; Фридман, Ян М .; и другие. (2009). «Массивно параллельное секвенирование: следующий большой шаг в генетической медицине». Американский журнал генетики человека. 85 (2): 142–54. Дои:10.1016 / j.ajhg.2009.06.022. ЧВК 2725244. PMID 19679224.
- ^ Торрес Э.М.; Williams, B.R .; Amon, A .; и другие. (2008). «Анеуплоидия: клетки, теряющие равновесие». Генетика. 179 (2): 737–46. Дои:10.1534 / genetics.108.090878. ЧВК 2429870. PMID 18558649.
- ^ Дикарь округ Колумбия (1977). «Микробная экология желудочно-кишечного тракта». Ежегодный обзор микробиологии. 31: 107–33. Дои:10.1146 / annurev.mi.31.100177.000543. PMID 334036.
- ^ Хамади М., Рыцарь Р. (2009). «Профилирование микробного сообщества для проектов по микробиому человека: инструменты, методы и проблемы». Геномные исследования. 19 (7): 1141–52. Дои:10.1101 / гр.085464.108. ЧВК 3776646. PMID 19383763.
