Циркулирующая опухолевая ДНК - Circulating tumor DNA
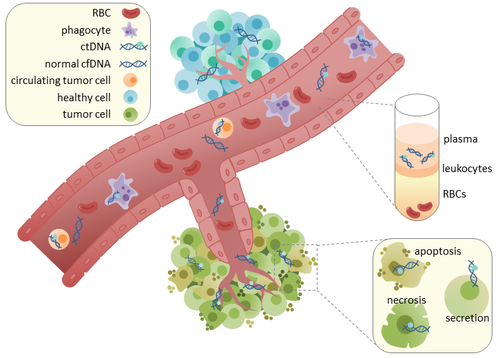
Циркулирующая опухолевая ДНК (ктДНК) опухоль -производные фрагментированные ДНК в кровотоке, который не связан с клетками. втДНК не следует путать с внеклеточной ДНК (вкДНК), более широким термином, который описывает ДНК, которая свободно циркулирует в кровотоке, но не обязательно имеет опухолевое происхождение. Поскольку ctDNA может отражать всю опухоль геном, он получил признание за его потенциальную клиническую полезность; «жидкие биопсии »В виде заборов крови можно брать в различные моменты времени для отслеживания прогрессирования опухоли на протяжении всего курса лечения.[1]
ктДНК происходит непосредственно из опухоли или из циркулирующие опухолевые клетки (ЦКО),[2] который описывает жизнеспособные, неповрежденные опухолевые клетки, которые отделяются от первичных опухолей и попадают в кровоток или лимфатический система. Точный механизм высвобождения ктДНК неясен. Биологические процессы, которые, как предполагается, участвуют в высвобождении ктДНК, включают: апоптоз и некроз из умирающих клеток или активное высвобождение из жизнеспособных опухолевых клеток.[3][4][5][6][7] Исследования на людях (здоровых и больных раком)[8] и ксенотрансплантаты мыши[9] показывают, что размер фрагментированной вкДНК составляет преимущественно 166 п.н., что соответствует длине ДНК, обернутой вокруг нуклеосома плюс компоновщик. Фрагментация такой длины может указывать на апоптотическая фрагментация ДНК, предполагая, что апоптоз может быть основным методом высвобождения ктДНК. Фрагментация вкДНК изменяется в плазме больных раком.[10][11]
В здоровые ткани проникает фагоциты несут ответственность за удаление апоптотических или некротических клеточных остатков, которые включают вкДНК.[12] Уровни cfDNA у здоровых пациентов присутствуют только на низких уровнях, но могут быть обнаружены более высокие уровни ctDNA у онкологических пациентов. Возможно, это происходит из-за неэффективной инфильтрации иммунных клеток к участкам опухоли, что снижает эффективный клиренс цтДНК из кровотока.[12] Сравнение мутаций в цДНК и ДНК, выделенных из первичных опухолей у одних и тех же пациентов, выявило наличие идентичных связанных с раком генетических изменений. [13][14] Это привело к возможности использования втДНК для более раннего выявления рака и последующего лечения. [15]
Методы
Преданалитические соображения
Когда кровь собирается в пробирки с ЭДТА и хранится, белые кровяные тельца начинают лизироваться и выделять геномную ДНК дикого типа в образец в количествах, которые обычно во много раз превышают содержание втДНК.[16] Это затрудняет обнаружение мутаций или других биомаркеров втДНК.[17] Использование имеющихся в продаже пробирок для стабилизации клеток может предотвратить или замедлить лизис лейкоцитов, тем самым уменьшая эффект разведения ктДНК.[18] Sherwood et al продемонстрировали превосходное обнаружение KRAS мутации в совпадающих образцах, собранных в пробирках с EDTA K3 и Streck BCT.[18] Преимущества пробирок для стабилизации клеток могут быть реализованы в ситуации, когда кровь не может быть немедленно преобразована в плазму.
Другие процедуры также могут уменьшить количество «загрязняющей» ДНК дикого типа и сделать обнаружение цтДНК более осуществимым:[18]
- Никогда не замораживайте образец крови перед извлечением плазмы для анализа на цДНК.
- Переведите образец в плазму в течение 2–4 часов (если собирали в пробирку с ЭДТА).
- Никогда не используйте гепаринизированные пробирки, гепарин подавляет ПЦР, имитируя спиральную структуру ДНК.
- Выполните этап двойного центрифугирования (центрифугируйте кровь для удаления плазмы, затем повторите с плазмой, чтобы удалить остатки на дне пробирки), чтобы удалить больше клеточного дебриса перед экстракцией ДНК.
- Плазма лучше сыворотки для восстановления цДНК[19]
Извлечение ктДНК
Основная привлекательность анализа втДНК заключается в том, что она извлекается неинвазивным способом путем сбора крови. Для получения вкДНК или втДНК обычно требуется собрать примерно 3 мл крови в EDTA трубы с покрытием. Использование ЭДТА важно для уменьшения свертывания крови. В плазма и сыворотка фракции крови могут быть отделены на стадии центрифугирования. впоследствии из этих фракций можно выделить цДНК или вкДНК. Хотя сыворотка, как правило, имеет более высокие уровни вкДНК, это в первую очередь связано с ДНК лимфоцитов.[20] Высокий уровень загрязнения вкДНК является субоптимальным, потому что это может снизить чувствительность обнаружения втДНК. Поэтому в большинстве исследований для выделения цДНК используется плазма. Затем плазму снова обрабатывают центрифугированием для удаления остаточных интактных клеток крови. Супернатант используется для экстракции ДНК, которую можно проводить с использованием имеющихся в продаже наборов.
Анализ ctDNA
Анализ ктДНК после экстракции требует использования различных методов амплификации и секвенирования. Эти методы можно разделить на две основные группы в зависимости от того, является ли цель опросом всех генов нецелевым подходом или целью является мониторинг определенных генов и мутаций с помощью целевого подхода.
Нецелевые подходы
Подходы к секвенированию всего генома или всего экзома могут быть необходимы для обнаружения новых мутаций в ДНК опухоли при мониторинге бремени болезни или отслеживании устойчивости к лекарствам.[21] Ненаправленные подходы также полезны в исследованиях для наблюдения за неоднородностью опухоли или для обнаружения новых мишеней для лекарств. Однако, хотя в некоторых приложениях могут потребоваться нецелевые методы, они более дороги и имеют более низкое разрешение. Это затрудняет обнаружение редких мутаций или в ситуациях, когда присутствуют низкие уровни ctDNA (например, минимальная остаточная болезнь). Кроме того, могут возникнуть проблемы с различением ДНК из опухолевых клеток и ДНК из нормальных клеток с использованием полногеномного подхода.
Для секвенирования всего генома или экзома обычно используют технологии высокопроизводительного секвенирования ДНК. Вместо этого ограничение секвенирования только всем экзомом может снизить расходы и увеличить скорость, но за счет потери информации о мутациях в некодирующих регуляторных областях ДНК.[22] Хотя простой просмотр полиморфизмов ДНК посредством секвенирования не позволяет отличить ДНК от опухолевых или нормальных клеток, эту проблему можно решить путем сравнения с контрольным образцом нормальной ДНК (например, ДНК, полученной с помощью буккальный мазок.) Важно отметить, что секвенирование всего генома и всего экзома полезно для первоначального обнаружения мутаций. Это дает информацию для использования более чувствительных целевых методов, которые затем можно использовать для мониторинга заболеваний.
Полногеномное секвенирование позволяет восстановить структурные свойства вкДНК, размер фрагментов и характер их фрагментации. Эти уникальные паттерны могут быть важным источником информации для улучшения обнаружения ctDNA или локализации ткани происхождения этих фрагментов.[23] Выбор размера коротких фрагментов (<150 п.н.) методами in vitro или in silico может улучшить восстановление мутаций и аберраций числа копий.[24]
Цифровое кариотипирование
Этот метод был первоначально разработан лабораторией Берт Фогельштейн, Луис Диас и Виктор Велкулеску в Университет Джона Хопкинса.[25] В отличие от обычного кариотипирование где краситель используется для окрашивания полос хромосом с целью визуализации хромосом, цифровое кариотипирование использует последовательности ДНК локусов по всему геному для расчета изменение количества копий.[25] Вариации числа копий распространены при раке и описывают ситуации, когда потеря гетерозиготности гена может привести к снижению функции из-за более низкой экспрессии или дупликации гена, что приводит к сверхэкспрессии.
Персонализированный анализ переставленных концов (PARE)
После секвенирования всего генома с использованием метода высокопроизводительного секвенирования, такого как Illumina HiSeq, к данным применяется PARE для анализа хромосомных перестроек и транслокаций. Изначально этот метод был разработан для анализа ДНК солидных опухолей, но был модифицирован для приложений стДНК.[25]
Метилирование ДНК и гидроксиметилирование
Правильный эпигенетический Маркировка важна для нормальной экспрессии генов и функции клеток, а аберрантные изменения в эпигенетических паттернах являются признаком рака.[26] Нормальный эпигенетический статус поддерживается в клетке, по крайней мере частично, через Метилирование ДНК.[27] Измерение паттернов аберрантного метилирования в ктДНК возможно благодаря стабильному метилированию участков ДНК, называемых «Острова CpG ». Метилирование ктДНК можно обнаружить через бисульфитная обработка. Обработка бисульфитом химически превращает неметилированные цитозины в урацил, оставляя метилированные цитозины неизмененными. Затем ДНК секвенируется, и можно идентифицировать любые изменения в структуре метилирования ДНК. Гидроксиметилирование ДНК является аналогичным ассоциированным маркером, который, как было показано, является прогностическим маркером здоровых и патологических состояний в вкДНК, включая рак. Измерение паттернов аберрантного гидроксиметилирования в ктДНК было доказано исследователями из Чикагского университета (лаборатория Чуан Хе,[28]) Стэнфордский университет (лаборатория Quake,[29]) и компания Кембридж Эпигенетикс.
Целевые подходы
При целевом подходе секвенирование ctDNA может быть направлено на генетическую панель, построенную на основе мутационных горячих точек для интересующего рака. Это особенно важно для информирования о лечении в ситуациях, когда мутации выявляются в мишенях, подверженных воздействию лекарств.[22] Персонализированный целевой анализ цтДНК для каждого пациента также возможен путем сочетания жидкой биопсии со стандартной первичной биопсией ткани. Секвенирование всего генома или всего экзома первичной биопсии опухоли позволяет обнаруживать генетические мутации, специфичные для опухоли пациента, и может использоваться для последующего целевого секвенирования втДНК пациента. Наивысшая чувствительность обнаружения цДНК достигается за счет целевого секвенирования конкретных однонуклеотидный полиморфизм (SNP). Обычно мутировавшие гены, такие как онкогены, которые обычно имеют горячие точки, являются хорошими кандидатами для целевых подходов к секвенированию. Напротив, большинство генов-супрессоров опухолей имеют широкий спектр возможных мутаций потери функции по всему гену и как таковые не подходят для целевого секвенирования.
Целевые подходы обладают преимуществом амплификации ктДНК через полимеразные цепные реакции (ПЦР) или цифровая ПЦР. Это особенно важно при анализе цтДНК не только из-за относительно низких уровней ДНК, циркулирующих в кровотоке, но также из-за того, что цтДНК составляет небольшую долю от всей экстрагированной внеклеточной ДНК.[22] Следовательно, амплификация интересующих областей может значительно улучшить чувствительность обнаружения цДНК. Однако амплификация с помощью ПЦР может привести к ошибкам, учитывая частоту ошибок, присущих ДНК-полимеразам. Ошибки, вносимые во время секвенирования, также могут снизить чувствительность обнаружения мутаций цДНК.
Капельная цифровая ПЦР (ddPCR)
Этот метод основан на цифровой ПЦР, первоначально названной Берт Фогельштейн Группа в Университет Джона Хопкинса. Droplet Digital PCR использует генератор капель для разделения отдельных частей ДНК на капли с использованием эмульсии масло / вода. Затем индивидуальная полимеразная цепная реакция происходит в каждой капле с использованием выбранных праймеров против областей ктДНК и продолжается до конечной точки. Присутствие представляющих интерес последовательностей измеряется флуоресцентными зондами, которые связываются с амплифицированной областью. ddPCR позволяет с высокой степенью количественной оценки частот аллелей и мутантов в ctDNA, но ограничивается количеством флуоресцентных зондов, которые можно использовать в одном анализе (до 5).[30] Чувствительность анализа может варьироваться в зависимости от количества проанализированной ДНК и составляет примерно 1 из 10 000.[30]
Бусины, эмульгирование, усиление и магнетизм (Луч )
Этот метод основан на Droplet Digital PCR, чтобы идентифицировать мутации в ctDNA с помощью проточной цитометрии.[31] После извлечения ктДНК из крови проводят ПЦР с праймерами, предназначенными для нацеливания на интересующие области. Эти праймеры также содержат определенные последовательности ДНК или метки. Амплифицированную ДНК смешивают с покрытыми стрептавидином магнитными шариками и эмульгируют в капли. Биотинилированные праймеры, предназначенные для связывания с метками, используются для амплификации ДНК. Биотинилирование позволяет амплифицированной ДНК связываться с магнитными шариками, которые покрыты стрептавидином. После завершения ПЦР гранулы, связанные с ДНК, отделяют с помощью магнита. Затем ДНК на гранулах денатурируют и дают возможность гибридизоваться с флуоресцентными олигонуклеотидами, специфичными для каждой матрицы ДНК. Затем полученные комплексы гранула-ДНК анализируют с помощью проточной цитометрии. Этот метод позволяет регистрировать частоты аллелей и мутаций из-за связывания с ddPCR. Однако, в отличие от ddPCR, можно опросить большее количество последовательностей ДНК из-за гибкости использования флуоресцентно связанных зондов. Еще одно преимущество этой системы состоит в том, что выделенную ДНК также можно использовать для последующего секвенирования.[32] Чувствительность составляет от 1,6 из 10 000 до 4,3 из 100 000.[30]
Персонализированное профилирование CAncer путем глубокого секвенирования (CAPP-Seq)
Этот метод был первоначально описан Аш Ализаде и группы Максимилиана Дина в Стэндфордский Университет. В этом методе используются биотинилированные олигонуклеотидные селекторные зонды для нацеливания последовательностей ДНК, имеющих отношение к обнаружению цДНК.[33] Общедоступные базы данных рака были использованы для создания библиотеки зондов против повторяющихся мутаций при раке путем расчета их индекса рецидива. Протокол был оптимизирован для низких уровней ДНК, наблюдаемых при сборе ктДНК. Затем выделенная ДНК подвергается глубокому секвенированию для повышения чувствительности. Этот метод позволяет исследовать сотни участков ДНК. Сообщается, что чувствительность обнаружения ктДНК CAPP-Seq составляет 2,5 молекулы на 1000000.[34]
Глубокое секвенирование меченых ампликонов (TAM-Seq)
TAM-Seq позволяет целенаправленно секвенировать целые гены для обнаружения мутаций в ктДНК.[35] Сначала выполняется общая стадия амплификации с использованием праймеров, которые охватывают весь интересующий ген на участках размером 150-200 пар оснований. Затем микрофлюидическая система используется для прикрепления адаптеров с уникальным идентификатором к каждому ампликону для дальнейшей амплификации ДНК в параллельных одноплексных реакциях. Было показано, что этот метод успешно идентифицирует мутации, разбросанные в гене-супрессоре опухоли TP53 у пациентов с запущенным раком яичников. Чувствительность этой техники - 1 из 50.
Безопасное секвенирование (Safe-Seq)
Этот метод был первоначально описан Берт Фогельштейн и его группа в Университет Джона Хопкинса. Safe-Seq снижает частоту ошибок при массовом параллельном секвенировании, чтобы повысить чувствительность к редким мутантам.[36] Это достигается путем добавления последовательности уникального идентификатора (UID) к каждой матрице ДНК. Затем ДНК амплифицируют с использованием добавленных UID и секвенируют. Все молекулы ДНК с одним и тем же UID (семейство UID) должны иметь одинаковую описанную последовательность ДНК, поскольку они были амплифицированы из одной молекулы. Однако мутации могут быть внесены путем амплификации, или неправильное назначение оснований может быть вызвано на этапах секвенирования и анализа. Наличие UID позволяет отделить эти методологические ошибки от истинных мутаций ктДНК. Мутация считается «супермутантом», если 95% секвенированных считываний совпадают. Чувствительность этого подхода составляет 9 из 1 миллиона.[30]
Дуплексное секвенирование
Этот метод является усовершенствованием единичных UID, добавленных в метод Safe-Seq.[37] При дуплексном секвенировании рандомизированная двухцепочечная ДНК действует как уникальные метки и прикрепляется к инвариантному спейсеру. Теги прикрепляются к обоим концам фрагмента ДНК (теги α и β), что приводит к созданию двух уникальных шаблонов для ПЦР - одной цепи с тегом α на 5 'конце и тега β на 3' конце и другой цепи. с тегом β на 5 'конце и тегом α на 3' конце. Затем эти фрагменты ДНК амплифицируют с праймерами против инвариантных последовательностей меток. Амплифицированная ДНК секвенируется и анализируется. ДНК с дуплексными адаптерами сравнивается, и мутации принимаются только при наличии консенсуса между обеими цепями. Этот метод учитывает как ошибки секвенирования, так и ошибки ранней стадии ПЦР-амплификации. Чувствительность подхода к обнаружению мутантов - 1 к 10 ^ 7.
CAPP-Seq с улучшенным интегрированным цифровым подавлением ошибок (iDES)
iDES улучшает анализ ктДНК с помощью CAPP-Seq, чтобы уменьшить количество ошибок и, следовательно, повысить чувствительность обнаружения.[34] Как сообщалось в 2016 году, iDES сочетает в себе CAPP-Seq с технологией последовательного дуплексного штрих-кодирования и вычислительным алгоритмом, который устраняет стереотипные ошибки, связанные с этапом гибридизации CAPP-Seq. Метод также включает дуплексное секвенирование, где это возможно, и включает методы более эффективного дуплексного восстановления из бесклеточной ДНК. Чувствительность этой улучшенной версии CAPP-Seq составляет 4 на 100 000 копий.
Соображения
«Нормальное» против обнаружения ДНК опухоли
Одна из проблем при использовании ктДНК в качестве биомаркера рака заключается в том, можно ли с помощью вкДНК отличить цтДНК от нормальных клеток. вкДНК выделяется доброкачественными клетками во время нормального клеточного обновления, но также и во время таких процедур, как хирургия, лучевая терапия, или же химиотерапия. Считается, что лейкоциты являются основными участниками вкДНК в сыворотке.[22]
Исследование
ктДНК в скрининге рака
Клиническая применимость ктДНК для обнаружения первичного заболевания частично ограничивается чувствительностью современной технологии; присутствуют низкие уровни ctDNA, и мутации драйвера неизвестны.[30]
ктДНК в мониторинге рака
Признаки заболевания традиционными методами визуализации, такими как CT, ДОМАШНИЙ ПИТОМЕЦ или же МРТ может отсутствовать после удаления опухоли. Таким образом, анализ ктДНК открывает потенциальные возможности для обнаружения минимальная остаточная болезнь (MRD) и, следовательно, возможность рецидива опухоли в случаях, когда объемная опухоль отсутствует обычными методами визуализации. Сравнение обнаружения MRD с помощью компьютерной томографии и ctDNA ранее проводилось у лиц с раком толстой кишки II стадии; В этом исследовании исследователи смогли обнаружить цТДНК у людей, у которых не было признаков клинической злокачественности с помощью компьютерной томографии, что позволяет предположить, что обнаружение цДНК имеет большую чувствительность для оценки МОБ.[22] Однако авторы признают, что анализ ктДНК не без ограничений; образцы плазмы, собранные после операции, могли предсказать рецидив через 36 месяцев только в 48% случаев.[22]
втДНК как прогностический биомаркер
Вопрос о том, можно ли использовать измерение количества или качества ктДНК для определения исходов у людей с раком, был предметом исследования. По состоянию на 2015 год это было очень неопределенным.[38] Хотя некоторые исследования показали тенденцию к более высоким уровням цДНК у людей с метастатическим раком на высокой стадии, бремя цтДНК не всегда коррелирует с традиционной стадией рака.[30] По состоянию на 2017 год казалось маловероятным, что ктДНК будет иметь клиническую ценность в качестве единственного предиктора прогноза.[39]
Исследования рака
Возникновение лекарственно-устойчивых опухолей из-за внутри- и межопухолевой гетерогенности является вопросом эффективности лечения. Незначительный генетический клон в опухоли может разрастаться после лечения, если он несет мутацию, устойчивую к лекарствам. При первоначальной биопсии эти клоны могут отсутствовать из-за низкой частоты или пространственного разделения клеток внутри опухоли. Например, поскольку при биопсии отбирается только небольшая часть опухоли, клоны, находящиеся в другом месте, могут остаться незамеченными. Это может ввести в заблуждение исследования, направленные на изучение роли неоднородности опухоли в прогрессировании и рецидиве рака. Использование ктДНК в исследованиях может облегчить эти опасения, поскольку может предоставить более репрезентативный «снимок экрана» генетического разнообразия рака как на первичных, так и на метастатических участках. Например, было показано, что ктДНК полезна при изучении клональной эволюции рака пациента до и после схем лечения.[40] Раннее выявление рака по-прежнему является сложной задачей, но недавний прогресс в анализе эпигенетических свойств вкДНК или разблокировки фрагментации повышает чувствительность жидкой биопсии.[41]
Проблемы реализации
Внедрение ктДНК в клиническую практику в значительной степени затруднено из-за отсутствия стандартизированных методов обработки и анализа ктДНК. Прежде чем анализ цтДНК может стать рутинным клиническим анализом, необходимо разработать стандартизацию методов сбора образцов (включая время сбора), последующей обработки (выделение и амплификация ДНК), количественной оценки и валидации. Кроме того, может потребоваться создание панели «стандартных» биомаркеров, ассоциированных с опухолью, с учетом разрешения текущих методов секвенирования и обнаружения ктДНК. Секвенирование опухолеспецифических аберраций в образцах плазмы также может помочь исключить контаминирующую вкДНК из анализа; повышенные уровни вкДНК из нормальных клеток могут быть связаны с причинами, не связанными с раком.[22]
Смотрите также
Рекомендации
- ^ Ван Дж., Масси С., Гарсия-Корбачо Дж., Мульер Ф., Брентон Дж., Калдас С., Пейси С., Бэрд Р., Розенфельд Н. (апрель 2017 г.). «Жидкие биопсии достигли совершеннолетия: к внедрению циркулирующей опухолевой ДНК». Обзоры природы Рак. 17 (4): 223–238. Дои:10.1038 / nrc.2017.7. PMID 28233803. S2CID 4561229.
- ^ Акча Х, Демирай А., Ярен А., Бир Ф, Коселер А., Ивакава Р., Багчи Дж., Йокота Дж. (Март 2013 г.). «Использование сывороточной ДНК и пиросеквенирования для обнаружения мутаций EGFR при немелкоклеточном раке легкого». Генетика рака. 206 (3): 73–80. Дои:10.1016 / j.cancergen.2013.01.005. PMID 23491080.
- ^ Шварценбах Х., Хун Д.С., Пантель К. (июнь 2011 г.). «Внеклеточные нуклеиновые кислоты как биомаркеры у онкологических больных». Обзоры природы. Рак. 11 (6): 426–37. Дои:10.1038 / nrc3066. PMID 21562580. S2CID 6061607.
- ^ Строун М., Анкер П. (июль 1972 г.). «Нуклеиновые кислоты, спонтанно выделяемые ушными раковинами живых лягушек». Биохимический журнал. 128 (3): 100П – 101П. Дои:10.1042 / bj1280100pb. ЧВК 1173871. PMID 4634816.
- ^ Строун М., Ляути Дж., Ледеррей С., Олсон-Санд А., Анкер П. (ноябрь 2001 г.). «О возможном происхождении и механизме апоптоза циркулирующей ДНК и активного высвобождения ДНК». Clinica Chimica Acta; Международный журнал клинической химии. 313 (1–2): 139–42. Дои:10.1016 / S0009-8981 (01) 00665-9. PMID 11694251.
- ^ Анкер П., Строун М., Морис П.А. (сентябрь 1975 г.). «Спонтанное высвобождение ДНК лимфоцитами крови человека, как показано в системе in vitro». Исследования рака. 35 (9): 2375–82. PMID 1149042.
- ^ Роджерс Дж. К., Болдт Д., Корнфельд С., Скиннер А., Валери С. Р. (июль 1972 г.). «Экскреция дезоксирибонуклеиновой кислоты лимфоцитами, стимулированными фитогемагглютинином или антигеном». Труды Национальной академии наук Соединенных Штатов Америки. 69 (7): 1685–9. Bibcode:1972ПНАС ... 69.1685Р. Дои:10.1073 / pnas.69.7.1685. ЧВК 426778. PMID 4505646.
- ^ Heitzer E, Auer M, Hoffmann EM, Pichler M, Gasch C, Ulz P, Lax S, Waldispuehl-Geigl J, Mauermann O, Mohan S, Pristauz G, Lackner C, Höfler G, Eisner F, Petru E, Sill H, Samonigg H, Pantel K, Riethdorf S, Bauernhofer T, Geigl JB, Speicher MR (июль 2013 г.). «Установление опухолеспецифических изменений количества копий в плазменной ДНК больных раком». Международный журнал рака. 133 (2): 346–56. Дои:10.1002 / ijc.28030. ЧВК 3708119. PMID 23319339.
- ^ Thierry AR, Mouliere F, Gongora C, Ollier J, Robert B., Ychou M, Del Rio M, Molina F (октябрь 2010 г.). «Происхождение и количественная оценка циркулирующей ДНК у мышей с ксенотрансплантатами колоректального рака человека». Исследования нуклеиновых кислот. 38 (18): 6159–75. Дои:10.1093 / nar / gkq421. ЧВК 2952865. PMID 20494973.
- ^ Mouliere F, Robert B., Arnau Peyrotte E, Del Rio M, Ychou M и др. (2011) Циркулирующая ДНК опухолевого происхождения характеризуется высокой степенью фрагментации. PLOS ONE 6 (9): e23418. https://doi.org/10.1371/journal.pone.0023418
- ^ Мульере Ф., Чандрананда Д., Пискорз А.М., Мур Е.К., Моррис Дж., Альборн Л.Б., Майр Р., Горанова Т., Марасс Ф., Хайдер К., Ван Дж.К.М., Супернат А, Худекова И., Гунарис И., Рос С., Хименес-Линан М., Гарсиа-Корбачо Дж., Патель К., Эструп О., Мерфи С., Элдридж, доктор медицины, Гейл Д., Стюарт Г. Д., Бердж Дж., Купер В. Н., Ван дер Хейден М. С., Мэсси К. Э., Уоттс К., Корри П., Пейси С., Бриндл К. М., Бэрд RD, Мау-Соренсен, Паркинсон, Калифорния, Смит, К.Г., Брентон, Дж. Д., Розенфельд Н. (2018). «Улучшенное обнаружение циркулирующей опухолевой ДНК с помощью анализа размера фрагмента». Sci Transl Med. 10 (466): eaat4921. Дои:10.1126 / scitranslmed.aat4921. ЧВК 6483061. PMID 30404863.CS1 maint: несколько имен: список авторов (связь)
- ^ а б Писецкий Д.С., Фэрхерст А.М. (июнь 2007 г.). «Происхождение внеклеточной ДНК во время очистки мертвых и умирающих клеток». Аутоиммунитет. 40 (4): 281–4. Дои:10.1080/08916930701358826. PMID 17516210. S2CID 11499768.
- ^ Васиухин В., Анкер П., Морис П., Ляути Дж., Ледеррей С., Строун М. (апрель 1994 г.). «Точечные мутации гена N-ras в ДНК плазмы крови пациентов с миелодиспластическим синдромом или острым миелолейкозом». Британский журнал гематологии. 86 (4): 774–779. Дои:10.1111 / j.1365-2141.1994.tb04828.x. PMID 7918071. S2CID 26365875.
- ^ Васиухин В., Строун М., Морис П., Ляути Дж., Ледеррей С., Анкер П. (май 1994 г.). «Точечные мутации K-ras в ДНК плазмы крови пациентов с колоректальными опухолями». Вызовы современной медицины: биотехнология сегодня. 5: 141–150.
- ^ Юн Э (июль 2014 г.). «Биомаркеры рака: Написано кровью». Природа. 511 (7511): 524–526. Bibcode:2014Натура.511..524Y. Дои:10.1038 / 511524a. PMID 25079538. S2CID 4445938.
- ^ Сюэ X, Тир, доктор медицины, Холен И., Чжу Ю.М., Уолл П.Дж. (июнь 2009 г.). «Оптимизация выхода и полезности циркулирующей внеклеточной ДНК из плазмы и сыворотки» (PDF). Clinica Chimica Acta; Международный журнал клинической химии. 404 (2): 100–4. Дои:10.1016 / j.cca.2009.02.018. PMID 19281804.
- ^ Нортон SE, Лехнер Дж. М., Уильямс Т., Фернандо М. Р. (октябрь 2013 г.). «Стабилизирующий реагент предотвращает загрязнение внеклеточной ДНК клеточной ДНК в плазме во время хранения и транспортировки образцов крови, как это определено с помощью цифровой ПЦР». Клиническая биохимия. 46 (15): 1561–5. Дои:10.1016 / j.clinbiochem.2013.06.002. PMID 23769817.
- ^ а б c Шервуд Дж. Л., Коркоран С., Браун Х, Шарп А. Д., Мусилова М., Кольманн А. (2016). «Оптимизированные преаналитические методы улучшают обнаружение мутаций KRAS в циркулирующей опухолевой ДНК (ктДНК) у пациентов с немелкоклеточным раком легкого (NSCLC)». PLOS ONE. 11 (2): e0150197. Bibcode:2016PLoSO..1150197S. Дои:10.1371 / journal.pone.0150197. ЧВК 4769175. PMID 26918901.
- ^ Валле А., Марк М., Бизьё А., Кури С.Э., Лакруа Н., Беннуна Дж., Дуйяр Д.Ю., Дени М.Г. (ноябрь 2013 г.). «Плазма является лучшим источником циркулирующей внеклеточной ДНК опухолевого происхождения, чем сыворотка, для обнаружения изменений EGFR у пациентов с опухолью легких». Рак легких. 82 (2): 373–4. Дои:10.1016 / j.lungcan.2013.08.014. PMID 24007628.
- ^ Ли TH, Montalvo L, Chrebtow V, Busch MP (февраль 2001 г.). «Количественное определение геномной ДНК в образцах плазмы и сыворотки: более высокие концентрации геномной ДНК в сыворотке, чем в плазме». Переливание. 41 (2): 276–82. Дои:10.1046 / j.1537-2995.2001.41020276.x. PMID 11239235. S2CID 45714834.
- ^ Цинь З., Любимов В.А., Чжоу Ц., Тонг Й., Лян Дж. (Апрель 2016 г.). «Внеклеточная циркулирующая опухолевая ДНК при раке». Китайский журнал рака. 35: 36. Дои:10.1186 / s40880-016-0092-4. ЧВК 4823888. PMID 27056366.
- ^ а б c d е ж грамм Heitzer E, Ulz P, Geigl JB (январь 2015 г.). «Циркулирующая опухолевая ДНК как жидкая биопсия рака». Клиническая химия. 61 (1): 112–23. Дои:10.1373 / Clinchem.2014.222679. PMID 25388429.
- ^ ван дер Поль Y, Mouliere F (2019). «На пути к раннему обнаружению рака путем расшифровки эпигенетических и экологических отпечатков бесклеточной ДНК». Раковая клетка. 36 (4): 350–368. Дои:10.1016 / j.ccell.2019.09.003. PMID 31614115.
- ^ Мульере Ф., Чандрананда Д., Пискорз А.М., Мур Е.К., Моррис Дж., Альборн Л.Б., Майр Р., Горанова Т., Марасс Ф., Хайдер К., Ван Дж.К.М., Супернат А, Худекова И., Гунарис И., Рос С., Хименес-Линан М., Гарсия-Корбачо Дж., Патель К., Эструп О., Мерфи С., Элдридж, доктор медицины, Гейл Д., Стюарт Г. Д., Бердж Дж., Купер В. Н., Ван дер Хейден М. С., Мэсси К. Э., Уоттс К., Корри П., Пейси С., Бриндл К. М., Бэрд RD, Мау-Соренсен, Паркинсон, Калифорния, Смит, К.Г., Брентон, Дж. Д., Розенфельд Н. (2018). «Улучшенное обнаружение циркулирующей опухолевой ДНК с помощью анализа размера фрагмента». Sci Transl Med. 10 (466): eaat4921. Дои:10.1126 / scitranslmed.aat4921. ЧВК 6483061. PMID 30404863.CS1 maint: несколько имен: список авторов (связь)
- ^ а б c Лири Р.Дж., Саусен М., Кинде И., Пападопулос Н., Карптен Д.Д., Крейг Д., О'Шонесси Дж., Кинзлер К.В., Пармиджани Дж., Фогельштейн Б., Диас Л.А., Велкулеску В.Е. (ноябрь 2012 г.). «Обнаружение хромосомных изменений в кровообращении онкологических больных с помощью полногеномного секвенирования». Научная трансляционная медицина. 4 (162): 162ra154. Дои:10.1126 / scitranslmed.3004742. ЧВК 3641759. PMID 23197571.
- ^ Преображенский Б.С. (1966). «[Современные перспективы и методика систематического лечения кохлеарного неврита и хронической лабиринтопатии]». Вестник Оториноларингологии (на русском). 28 (1): 3–11. PMID 5988180.
- ^ Бомонт Г., Доббинс С., Латта Д., Макмиллин В.П. (май 1990 г.). «Меквитазин в лечении сенной лихорадки». Британский журнал клинической практики. 44 (5): 183–8. PMID 1975200.
- ^ Ли В, Чжан Икс, Лу Х, Ю Л, Сонг И, Ло Зи и др. (Октябрь 2017 г.). «Сигнатуры 5-гидроксиметилцитозина в циркулирующей внеклеточной ДНК в качестве диагностических биомаркеров рака человека». Клеточные исследования. 27 (10): 1243–1257. Дои:10.1038 / cr.2017.121. ЧВК 5630683. PMID 28925386.
- ^ Song CX, Yin S, Ma L, Wheeler A, Chen Y, Zhang Y, Liu B, Xiong J, Zhang W, Hu J, Zhou Z, Dong B, Tian Z, Jeffrey SS, Chua MS, So S, Li W , Вэй Й, Дяо Дж., Се Д., Quake SR (октябрь 2017 г.). «Сигнатуры 5-гидроксиметилцитозина в внеклеточной ДНК предоставляют информацию о типах и стадиях опухоли». Клеточные исследования. 27 (10): 1231–1242. Дои:10.1038 / cr.2017.106. ЧВК 5630676. PMID 28820176.
- ^ а б c d е ж Butler TM, Spellman PT, Gray J (февраль 2017 г.). «Циркулирующая опухолевая ДНК как инструмент раннего обнаружения и диагностики». Текущее мнение в области генетики и развития. 42: 14–21. Дои:10.1016 / j.gde.2016.12.003. PMID 28126649.
- ^ Dressman D, Yan H, Traverso G, Kinzler KW, Vogelstein B (июль 2003 г.). «Преобразование отдельных молекул ДНК в флуоресцентные магнитные частицы для обнаружения и подсчета генетических вариаций». Труды Национальной академии наук Соединенных Штатов Америки. 100 (15): 8817–22. Bibcode:2003ПНАС..100.8817Д. Дои:10.1073 / pnas.1133470100. ЧВК 166396. PMID 12857956.
- ^ Диль Ф., Ли М., Хе И, Кинзлер К. В., Фогельштейн Б., Дрессман Д. (июль 2006 г.). «BEAMing: одномолекулярная ПЦР на микрочастицах в эмульсиях вода-в-масле». Природные методы. 3 (7): 551–9. Дои:10.1038 / nmeth898. PMID 16791214. S2CID 7059151.
- ^ Ньюман А.М., Братман С.В., То Дж., Винн Дж. Ф., Эклов Н. С., Модлин Л. А., Лю С. Л., Нил Дж. В., Уэйкли Х.А., Мерритт Р. Э., Шрагер Дж. Б., Лу Б. В., Ализаде А. А., Дин М. (май 2014 г.). «Сверхчувствительный метод количественного определения циркулирующей опухолевой ДНК с широким охватом пациентов». Природа Медицина. 20 (5): 548–54. Дои:10,1038 / нм.3519. ЧВК 4016134. PMID 24705333.
- ^ а б Ньюман А.М., Лавджой А.Ф., Класс Д.М., Курц Д.М., Чабон Дж. Дж., Шерер Ф. и др. (Май 2016). «Встроенное подавление цифровых ошибок для улучшенного обнаружения циркулирующей ДНК опухоли». Природа Биотехнологии. 34 (5): 547–555. Дои:10.1038 / nbt.3520. ЧВК 4907374. PMID 27018799.
- ^ Форшью Т., Муртаза М., Паркинсон С., Гейл Д., Цуй Д.В., Капер Ф, Доусон С.Дж., Пискорз А.М., Хименес-Линан М., Бентли Д., Хадфилд Дж., Мэй А.П., Калдас С., Брентон Д.Д., Розенфельд Н. (май 2012 г.) . «Неинвазивная идентификация и мониторинг раковых мутаций с помощью целевого глубокого секвенирования ДНК плазмы». Научная трансляционная медицина. 4 (136): 136ra68. Дои:10.1126 / scitranslmed.3003726. PMID 22649089. S2CID 34723244.
- ^ Kinde I, Wu J, Papadopoulos N, Kinzler KW, Vogelstein B (июнь 2011 г.). «Обнаружение и количественная оценка редких мутаций с массовым параллельным секвенированием». Труды Национальной академии наук Соединенных Штатов Америки. 108 (23): 9530–5. Bibcode:2011ПНАС..108.95 30К. Дои:10.1073 / pnas.1105422108. ЧВК 3111315. PMID 21586637.
- ^ Kennedy SR, Schmitt MW, Fox EJ, Kohrn BF, Salk JJ, Ahn EH, Prindle MJ, Kuong KJ, Shen JC, Risques RA, Loeb LA (ноябрь 2014 г.). «Обнаружение сверхнизкочастотных мутаций с помощью дуплексного секвенирования». Протоколы природы. 9 (11): 2586–606. Дои:10.1038 / nprot.2014.170. ЧВК 4271547. PMID 25299156.
- ^ Rapisuwon S, Vietsch EE, Wellstein A (2016). «Циркулирующие биомаркеры для наблюдения за прогрессированием и лечением рака». Журнал вычислительной и структурной биотехнологии. 14: 211–22. Дои:10.1016 / j.csbj.2016.05.004. ЧВК 4913179. PMID 27358717.
- ^ Кроули Э, Ди Николантонио Ф, Лупакис Ф, Барделли А (август 2013 г.). «Жидкая биопсия: мониторинг генетики рака в крови». Обзоры природы. Клиническая онкология. 10 (8): 472–84. Дои:10.1038 / nrclinonc.2013.110. PMID 23836314. S2CID 25537784.
- ^ Murtaza M, Dawson SJ, Pogrebniak K, Rueda OM, Provenzano E, Grant J, Chin SF, Tsui DW, Marass F, Gale D, Ali HR, Shah P, Contente-Cuomo T, Farahani H, Shumansky K, Kingsbury Z, Хамфрей С., Бентли Д., Шах С.П., Уоллис М., Розенфельд Н., Калдас С. (ноябрь 2015 г.). «Мультифокальная клональная эволюция с использованием циркулирующей опухолевой ДНК в случае метастатического рака груди». Nature Communications. 6: 8760. Bibcode:2015НатКо ... 6,8760 млн. Дои:10.1038 / ncomms9760. ЧВК 4659935. PMID 26530965.
- ^ ван дер Поль Y, Mouliere F (2019). «На пути к раннему обнаружению рака путем расшифровки эпигенетических и экологических отпечатков бесклеточной ДНК». Раковая клетка. 36 (4): 350–368. Дои:10.1016 / j.ccell.2019.09.003. PMID 31614115.
дальнейшее чтение
- Применение метилирования циркулирующей опухолевой ДНК в диагностике рака легких Май 2019
- Циркулирующая ДНК опухоли: новое поколение биомаркеров рака Февраль 2014 г.
- ctDNA "Жидкая биопсия" может революционизировать лечение рака Ноя 2014
- Ньюман А.М., Братман С.В., То Дж., Винн Дж. Ф., Эклов Н. С., Модлин Л. А., Лю С. Л., Нил Дж. В., Уэйкли Х.А., Мерритт Р. Э., Шрагер Дж. Б., Лу Б. В., Ализаде А. А., Дин М. (май 2014 г.). «Сверхчувствительный метод количественного определения циркулирующей опухолевой ДНК с широким охватом пациентов». Природа Медицина. 20 (5): 548–54. Дои:10,1038 / нм.3519. ЧВК 4016134. PMID 24705333.
- Карачалиу Н., Майо-де-Лас-Касас К., Молина-Вила, Массачусетс, Розелл Р. (март 2015 г.). «Жидкая биопсия в реальном времени становится реальностью в лечении рака». Анналы трансляционной медицины. 3 (3): 36. Дои:10.3978 / j.issn.2305-5839.2015.01.16. ЧВК 4356857. PMID 25815297.
- Марусина, Катя (8 февраля 2018). «Выявление циркулирующей ДНК опухоли». ClinicalOMICs. Получено 5 марта 2018.
- Дю-Буа, Асанте (2019). «Жидкая биопсия при раке яичников с использованием циркулирующих опухолевых ДНК и клеток: готовы к прайм-тайму?». Письма о раке. 468: 59–71. Дои:10.1016 / j.canlet.2019.10.014. PMID 31610267.