Полимеразная цепная реакция в реальном времени - Real-time polymerase chain reaction
Эта статья слишком полагается на Рекомендации к основные источники. (Июль 2018 г.) (Узнайте, как и когда удалить этот шаблон сообщения) |
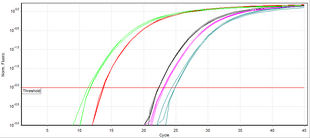
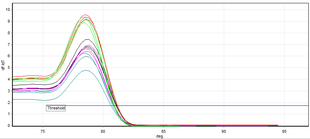
А полимеразная цепная реакция в реальном времени (ПЦР в реальном времени), также известный как количественная полимеразная цепная реакция (КПЦР), это лабораторная техника из молекулярная биология на основе полимеразной цепной реакции (ПЦР). Он контролирует усиление целевого ДНК молекулы во время ПЦР (т.е.в реальном времени), а не на ее конце, как в обычной ПЦР. ПЦР в реальном времени может использоваться как количественно (количественная ПЦР в реальном времени), так и полуколичественно (то есть выше / ниже определенного количества молекул ДНК) (полуколичественная ПЦР в реальном времени).
Два общих метода обнаружения продуктов ПЦР в ПЦР в реальном времени: (1) неспецифический флуоресцентные красители который вставлять с любой двухцепочечной ДНК и (2) специфичной для последовательности ДНК-зонды состоящий из олигонуклеотиды которые помечены флуоресцентный репортер, который разрешает обнаружение только после гибридизация зонда с его комплементарной последовательностью.
В Минимум информации для публикации количественных экспериментов ПЦР в реальном времени (MIQE) предлагает сокращение КПЦР использоваться для количественной ПЦР в реальном времени и что ОТ-КПЦР использоваться для обратной транскрипции - КПЦР.[1] Акроним «ОТ-ПЦР» обычно означает полимеразная цепная реакция с обратной транскрипцией и не ПЦР в реальном времени, но не все авторы придерживаются этого соглашения.[2]
Фон
Клетки всех организмов регулируют экспрессия гена по обороту транскриптов генов (одноцепочечные РНК ): Количество экспрессируемого гена в клетке можно измерить количеством копий транскрипта РНК этого гена, присутствующего в образце. Для надежного обнаружения и количественной оценки экспрессии гена из небольших количеств РНК необходима амплификация транскрипта гена. В полимеразной цепной реакции (ПЦР) - распространенный метод амплификации ДНК; для ПЦР на основе РНК образец РНК сначала подвергается обратной транскрипции в комплементарная ДНК (кДНК) с обратная транскриптаза.
Для амплификации небольших количеств ДНК используется та же методология, что и в обычной ПЦР с использованием матрицы ДНК, по крайней мере, одной пары специфических грунтовки, дезоксирибонуклеотиды, подходящий буферный раствор и термостабильный ДНК-полимераза. Вещество, отмеченное знаком флуорофор добавляется к этой смеси в термоциклер который содержит датчики для измерения флуоресценция флуорофора после его возбуждения при требуемом длина волны позволяя измерить скорость генерации для одного или нескольких конкретных продуктов. Это позволяет измерять скорость образования амплифицированного продукта в каждом цикле ПЦР. Полученные таким образом данные могут быть проанализированы компьютерным программным обеспечением для расчета относительная экспрессия гена (или же количество копий мРНК) в нескольких образцах. Количественная ПЦР также может применяться для обнаружения и количественного определения ДНК в образцах для определения присутствия и количества конкретной последовательности ДНК в этих образцах.[3] Это измерение выполняется после каждого цикла амплификации, и поэтому этот метод называется ПЦР в реальном времени (то есть немедленной или одновременной ПЦР). В случае количественного определения РНК матрицей является комплементарная ДНК (кДНК), которая получена обратная транскрипция из рибонуклеиновая кислота (РНК). В этом случае используется метод количественной RT-PCR или Q-RT-PCR.
Количественная ПЦР и Микрочип ДНК современные методики обучения экспрессия гена. Для измерения количества мРНК использовались более старые методы: Дифференциальный дисплей, Анализ защиты от РНКазы и северное пятно. Нозерн-блоттинг часто используется для оценки уровня экспрессии гена путем визуализации количества его транскрипта мРНК в образце. В этом методе очищенная РНК разделяется электрофорез в агарозном геле, переносится на твердую матрицу (например, нейлоновую мембрану) и исследуется специальным Зонд ДНК или РНК то есть дополнительный интересующему гену. Хотя этот метод все еще используется для оценки экспрессии генов, он требует относительно большого количества РНК и предоставляет только качественную или полуколичественную информацию об уровнях мРНК.[4] Ошибки оценки, возникающие из-за вариаций в методе количественной оценки, могут быть результатом целостности ДНК, эффективность ферментов и многие другие факторы. По этой причине ряд стандартизация системы (часто называемые методы нормализации ) были разработаны. Некоторые из них были разработаны для количественной оценки общей экспрессии гена, но наиболее распространенные нацелены на количественную оценку конкретного изучаемого гена по отношению к другому гену, называемому нормализующим геном, который выбран по почти постоянному уровню экспрессии. Эти гены часто выбираются из гены домашнего хозяйства поскольку их функции связаны с основными сотовый выживание обычно подразумевает конститутивная экспрессия гена.[5][6] Это позволяет исследователям сообщать соотношение экспрессии интересующих генов, деленное на экспрессию выбранного нормализатора, тем самым позволяя сравнивать первый, фактически не зная его абсолютного уровня экспрессии.
Наиболее часто используемые нормализующие гены - это те, которые кодируют следующие молекулы: тубулин, глицеральдегид-3-фосфатдегидрогеназа, альбумин, циклофилин, и рибосомные РНК.[4]
Основные принципы
ПЦР в реальном времени проводится в термоциклер с возможностью освещать каждый образец лучом света по крайней мере одной указанной длины волны и обнаруживать флуоресценцию, испускаемую возбужденным флуорофор. Термоциклер также может быстро нагревать и охлаждать образцы, тем самым используя физико-химические свойства нуклеиновые кислоты и ДНК-полимераза.
Процесс ПЦР обычно состоит из серии изменений температуры, которые повторяются 25–50 раз. Эти циклы обычно состоят из трех стадий: первая, при температуре около 95 ° C, позволяет разделить двойную цепь нуклеиновой кислоты; второй, при температуре около 50–60 ° C, позволяет связывать праймеры с матрицей ДНК;[7] третий, при температуре 68–72 ° C, способствует полимеризация осуществляется ДНК-полимеразой. Из-за небольшого размера фрагментов последний этап обычно пропускается в этом типе ПЦР, поскольку фермент может увеличивать их количество во время перехода между этапом выравнивания и этапом денатурирования. Кроме того, в четырехэтапной ПЦР флуоресценция измеряется во время коротких температурных фаз, продолжающихся всего несколько секунд в каждом цикле, при температуре, например, 80 ° C, чтобы уменьшить сигнал, вызванный присутствием димеров праймеров. когда используется неспецифический краситель.[8] Температура и время, используемые для каждого цикла, зависят от широкого спектра параметров, таких как: фермент, используемый для синтеза ДНК, концентрация двухвалентных ионов и дезоксирибонуклеотиды (dNTP) в реакции и температуре связывания праймеров.[9]час
Химическая классификация
Методики ПЦР в реальном времени можно классифицировать по химическому составу, используемому для обнаружения продукта ПЦР, специфических или неспецифических флуорохромов.
Неспецифическое обнаружение: ПЦР в реальном времени с двухцепочечными ДНК-связывающими красителями в качестве репортеров
ДНК-связывающий краситель связывается со всеми двухцепочечными (ds) ДНК в ПЦР, увеличивая квантовый выход флуоресценции красителя. Поэтому увеличение количества продукта ДНК во время ПЦР приводит к увеличению интенсивности флуоресценции, измеряемой в каждом цикле. Однако красители дцДНК, такие как SYBR Зеленый будет связываться со всеми продуктами ПЦР дцДНК, включая неспецифические продукты ПЦР (такие как Праймер димер ). Это потенциально может помешать или помешать точному мониторингу намеченной целевой последовательности.
В ПЦР в реальном времени с красителями дцДНК реакция готовится как обычно, с добавлением флуоресцентного красителя дцДНК. Затем реакция запускается в прибор для ПЦР в реальном времени, а после каждого цикла интенсивность флуоресценции измеряется детектором; краситель флуоресцирует только при связывании с дцДНК (т.е. с продуктом ПЦР). Преимущество этого метода состоит в том, что для проведения амплификации требуется только пара праймеров, что снижает затраты; несколько последовательностей-мишеней можно контролировать в пробирке с использованием различных типов красителей.
Специфическое обнаружение: метод флуоресцентного репортерного зонда

Флуоресцентный репортерные зонды обнаруживают только ДНК, содержащую последовательность, комплементарную зонду; поэтому использование репортерного зонда значительно увеличивает специфичность и позволяет выполнять методику даже в присутствии другой дцДНК. Используя разноцветные метки, флуоресцентные зонды можно использовать в мультиплексных анализах для мониторинга нескольких целевых последовательностей в одной пробирке. Специфичность флуоресцентных репортерных зондов также предотвращает вмешательство в измерения, вызванное димеры праймеров, которые являются нежелательными потенциальными побочными продуктами в ПЦР. Однако флуоресцентные репортерные зонды не предотвращают ингибирующее действие димеров праймеров, которое может подавлять накопление желаемых продуктов в реакции.
В этом методе используется зонд на основе ДНК с флуоресцентным репортером на одном конце и тушитель флуоресценции на противоположном конце зонда. Непосредственная близость репортера к тушителю предотвращает обнаружение его флуоресценции; пробой зонда от 5 'до 3' экзонуклеаза деятельность Полимераза Taq нарушает близость репортер-тушитель и, таким образом, допускает непогашенную флуоресценцию, которую можно обнаружить после возбуждение с лазером. Таким образом, увеличение количества продукта, нацеленного репортерным зондом, в каждом цикле ПЦР вызывает пропорциональное увеличение флуоресценции из-за разрушения зонда и высвобождения репортера.
- ПЦР готовится как обычно (см. ПЦР ), и добавляется зонд-репортер.
- Когда реакция начинается, во время отжиг На этапе ПЦР и зонд, и праймеры отжигаются с ДНК-мишенью.
- Полимеризация новой цепи ДНК инициируется праймерами, и как только полимераза достигает зонда, ее 5'-3'-экзонуклеаза разрушает зонд, физически отделяя флуоресцентный репортер от гасителя, что приводит к увеличению флуоресценции.
- Флуоресценция обнаруживается и измеряется в машине для ПЦР в реальном времени, и ее геометрическое увеличение, соответствующее экспоненциальному увеличению продукта, используется для определения цикла количественного определения. (Cq) в каждой реакции.
Анализ температуры плавления

ПЦР в реальном времени позволяет идентифицировать конкретные амплифицированные фрагменты ДНК с помощью анализа их температура плавления (также называемый Тм значение, от мelting ттемпература). Используемый метод обычно представляет собой ПЦР с двухцепочечными ДНК-связывающими красителями в качестве репортеров, а используемый краситель - обычно SYBR Green. Температура плавления ДНК специфична для амплифицированного фрагмента. Результаты этого метода получены путем сравнения кривых диссоциации проанализированных образцов ДНК.[11]
В отличие от обычной ПЦР, этот метод позволяет избежать предыдущего использования электрофорез методы для демонстрации результатов всех образцов. Это потому, что, несмотря на то, что это кинетический метод, количественная ПЦР обычно оценивается в определенной конечной точке. Поэтому метод обычно обеспечивает более быстрые результаты и / или использует меньше реагентов, чем электрофорез. Если требуется последующий электрофорез, необходимо протестировать только те образцы, которые оказались сомнительными при ПЦР в реальном времени, и / или утвердить результаты для образцов, которые дали положительный результат на определенный детерминант.
Моделирование
В отличие от ПЦР по конечной точке (обычной ПЦР), ПЦР в реальном времени позволяет контролировать желаемый продукт в любой момент процесса амплификации путем измерения флуоресценции (в реальном масштабе времени измеряется ее уровень выше заданного порога). Обычно используемый метод количественного определения ДНК с помощью ПЦР в реальном времени основан на построении графика зависимости флуоресценции от количества циклов на логарифмическая шкала. Порог обнаружения флуоресценции на основе ДНК устанавливается в 3-5 раз больше стандартное отклонение сигнала шума над фоном. Количество циклов, при которых флуоресценция превышает порог, называется пороговым циклом (Cт) или, согласно рекомендациям MIQE, цикл количественной оценки (Cq).[12]
Во время фазы экспоненциальной амплификации количество целевой ДНК-матрицы (ампликона) удваивается каждый цикл. Например, образец ДНК, у которого Cq предшествует таковому у другого образца на 3 цикла, содержащих 23 = В 8 раз больше шаблона. Однако эффективность амплификации часто варьируется среди праймеров и матриц. Следовательно, эффективность комбинации праймер-матрица оценивается в титрование поэкспериментируйте с серийными разведениями ДНК-матрицы для создания стандартная кривая изменения в (Cq) при каждом разведении. В склон из линейная регрессия затем используется для определения эффективности амплификации, которая составляет 100%, если разведение 1: 2 приводит к (Cq) разница в 1. Метод порогового цикла делает несколько предположений о механизме реакции и полагается на данные из областей с низким отношением сигнал-шум профиля усиления, которые могут вносить существенные различия во время анализа данных.[13]
Для количественной оценки экспрессии генов (Cq) для РНК или ДНК из интересующего гена вычитается из (Cq) РНК / ДНК из гена домашнего хозяйства в одном образце, чтобы нормализовать различия в количестве и качестве РНК между разными образцами. Эта процедура нормализации обычно называется ΔCт-метод[14] и позволяет сравнивать экспрессию интересующего гена среди различных образцов. Однако для такого сравнения экспрессия нормализующего эталонного гена должна быть очень похожей во всех образцах. Поэтому выбор эталонного гена, удовлетворяющего этому критерию, очень важен и часто является сложной задачей, потому что лишь очень немногие гены демонстрируют одинаковые уровни экспрессии в различных условиях или тканях.[15][16] Хотя анализ пороговых значений цикла интегрирован со многими коммерческими программными системами, существуют более точные и надежные методы анализа данных профиля амплификации, которые следует учитывать в случаях, когда воспроизводимость является проблемой.[13]
Также были предложены методы количественной оценки количественной ПЦР на основе механизмов, и их преимущество состоит в том, что они не требуют стандартной кривой для количественной оценки. Такие методы, как MAK2[17] было показано, что они имеют такие же или лучшие количественные характеристики по сравнению с методами стандартной кривой. Эти основанные на механизмах методы используют знания о процессе амплификации полимеразы для получения оценок исходной концентрации образца. Расширение этого подхода включает в себя точную модель всего профиля реакции ПЦР, которая позволяет использовать данные с высоким соотношением сигнал-шум и возможность проверки качества данных перед анализом.[13]
Согласно исследованию Ruijter et al.[18] MAK2 предполагает постоянную эффективность амплификации во время реакции ПЦР. Однако теоретический анализ полимеразной цепной реакции, из которой был получен MAK2, показал, что эффективность амплификации не является постоянной во время ПЦР. В то время как количественная оценка MAK2 обеспечивает надежные оценки концентрации целевой ДНК в образце при нормальных условиях кПЦР, MAK2 не дает надежной количественной оценки целевой концентрации для анализов количественной ПЦР с помощью конкурентов.
Приложения
Существует множество применений количественной полимеразной цепной реакции в лаборатория. Обычно он используется как для диагностический и фундаментальные исследования. Использование этого метода в промышленности включает количественную оценку микробной нагрузки в пищевых продуктах или растительных веществах, обнаружение ГМО (Генетически модифицированные организмы ), а также количественная оценка и генотипирование вирусных патогенов человека.
Количественная оценка экспрессии генов
Количественная оценка экспрессии генов с помощью традиционных методов обнаружения ДНК ненадежна. Обнаружение мРНК на северное пятно или продукты ПЦР на гель или же Саузерн-блот не позволяет точного количественного определения.[19] Например, за 20–40 циклов типичной ПЦР количество продукта ДНК достигает плато это не коррелирует напрямую с количеством целевой ДНК в начальной ПЦР.[20]
ПЦР в реальном времени может использоваться для количественной оценки нуклеиновые кислоты двумя обычными методами: относительной количественной и абсолютной количественной оценкой.[21] Абсолютное количественное определение дает точное количество молекул-мишеней ДНК по сравнению со стандартами ДНК с использованием калибровочная кривая. Поэтому важно, чтобы результаты ПЦР образца и стандарта были одинаковыми. эффективность усиления.[22]Относительная количественная оценка основана на внутренних эталонных генах для определения кратных различий в экспрессии целевого гена. Количественная оценка выражается как изменение уровней экспрессии мРНК, интерпретируемое как комплементарная ДНК (кДНК, генерируемая обратная транскрипция мРНК). Относительную количественную оценку проводить легче, поскольку она не требует калибровочной кривой, поскольку количество исследуемого гена сравнивается с количеством контрольного эталонного гена.
Поскольку единицы, используемые для выражения результатов относительной количественной оценки, не важны, результаты можно сравнивать по ряду различных RTqPCR. Причина использования одного или нескольких генов домашнего хозяйства состоит в том, чтобы исправить неспецифические вариации, такие как различия в количестве и качестве используемой РНК, которые могут повлиять на эффективность обратной транскрипции и, следовательно, на эффективность всего процесса ПЦР. Однако наиболее важным аспектом процесса является то, что эталонный ген должен быть стабильным.[23]
Отбор этих референсных генов традиционно проводился в молекулярная биология с использованием качественных или полуколичественных исследований, таких как визуальный осмотр гелей РНК, нозерн-блоттинг денситометрия или полуколичественная ПЦР (имитация ПЦР). Теперь в геном эры, можно провести более подробную оценку многих организмов, используя транскриптомные технологии.[24] Однако исследования показали, что амплификация большинства эталонных генов, используемых для количественной оценки экспрессии мРНК, варьируется в зависимости от экспериментальных условий.[25][26][27] Поэтому необходимо провести первоначальную статистически тщательное методологическое исследование с целью выбора наиболее подходящего референсного гена.
Был разработан ряд статистических алгоритмов, которые могут определять, какой ген или гены наиболее подходят для использования в данных условиях. Такие, как geNORM или BestKeeper, могут сравнивать пары или геометрические средства для матрицы различных референсных генов и ткани.[28][29]
Диагностическое использование
Качественная диагностическая ПЦР применяется для быстрого обнаружения нуклеиновые кислоты которые являются диагностическими, например, инфекционные заболевания, рак и генетические аномалии. Внедрение качественных ПЦР-анализов в лаборатория клинической микробиологии значительно улучшила диагностику инфекционных заболеваний,[30] и используется в качестве инструмента для обнаружения новых болезней, таких как новые штаммы грипп и коронавирус,[31] в диагностические тесты.[32][33]
Микробиологические применения
Количественная ПЦР также используется микробиологами, работающими в области безопасности пищевых продуктов, их порчи и ферментации, а также для оценки микробного риска качества воды (питьевая и рекреационная вода), а также в сфере охраны здоровья населения.[34]
qPCR может также использоваться для амплификации таксономических или функциональных маркеров генов в ДНК, взятых из образцов окружающей среды.[35] Маркеры представлены генетическими фрагментами ДНК или комплементарной ДНК.[35] Путем амплификации определенного мягкого элемента можно количественно определить количество элемента в образце до амплификации.[35] Использование таксономических маркеров (рибосомных генов) и количественной ПЦР может помочь определить количество микроорганизмов в образце и определить различные семейства, роды или виды на основе специфичности маркера.[35] Использование функциональных маркеров (генов, кодирующих белок) может показать экспрессию генов в сообществе, что может раскрыть информацию об окружающей среде.[35]
Обнаружение фитопатогенов
Сельскохозяйственная промышленность постоянно стремится производить отростки или саженцы растений, свободные от патогенных микроорганизмов, чтобы предотвратить экономические потери и сохранить здоровье. Были разработаны системы, позволяющие обнаруживать небольшие количества ДНК Phytophthora ramorum, оомицет, убивающий Дубы и другие виды, смешанные с ДНК растения-хозяина. Различение ДНК патогена и растения основано на амплификации ITS-последовательностей, спейсеров, расположенных в рибосомная РНК области кодирования гена, характерные для каждого таксона.[36] Полевые версии этого метода также были разработаны для идентификации того же патогена.[37]
Обнаружение генетически модифицированных организмов
qPCR с использованием обратной транскрипции (RT-qPCR) может использоваться для обнаружения ГМО учитывая его чувствительность и динамический диапазон при обнаружении ДНК. Альтернативы, такие как анализ ДНК или белка, обычно менее чувствительны. Используются специфические праймеры, которые амплифицируют не трансген, а промоутер, терминатор или даже промежуточные последовательности, используемые в процессе создания вектора. Поскольку процесс создания трансгенного растения обычно приводит к встраиванию более чем одной копии трансгена, его количество также обычно оценивается. Это часто выполняется путем относительной количественной оценки с использованием контрольного гена из обработанных видов, который присутствует только в виде единственной копии.[38][39]
Клиническая количественная оценка и генотипирование
Вирусы могут присутствовать у людей из-за прямого заражения или сопутствующих инфекций, которые вызывают диагноз сложно использовать классические техники и может привести к неправильному прогноз и лечение. Использование количественной ПЦР позволяет как количественную оценку, так и генотипирование (характеристику штамма, выполняемую с использованием кривых плавления) вируса, такого как Вирус гепатита В.[40] Степень заражения, определяемая количественно как количество копий вирусного генома на единицу ткани пациента, во многих случаях имеет значение; например, вероятность того, что тип 1 Вирус простого герпеса reactivates связано с количеством зараженных нейроны в ганглии.[41] Эта количественная оценка проводится либо с обратной транскрипцией, либо без нее, как это происходит, если вирус интегрируется в геном человека в любой момент его цикла, например, в случае ВПЧ (вирус папилломы человека), где некоторые его варианты связаны с появлением рак шейки матки.[42]
Рекомендации
- ^ Бастин С.А., Бенес В., Гарсон Дж., Хеллеманс Дж., Хаггетт Дж., Кубиста М., Мюллер Р., Нолан Т., Пфаффл М.В., Шипли Г.Л., Вандесомпеле Дж., Виттвер Коннектикут (2009). «Рекомендации MIQE: минимум информации для публикации количественных экспериментов ПЦР в реальном времени». Клиническая химия. 55 (4): 611–622. Дои:10.1373 / Clinchem.2008.112797. PMID 19246619.
- ^ Логан, Джули; Эдвардс, Кирстин и Сондерс, Ник, ред. (2009). ПЦР в реальном времени: современные технологии и приложения. Caister Academic Press. ISBN 978-1-904455-39-4.
- ^ Уотсон, Дж. Д.; Бейкер, Т. А; Белл, S P; Ганн, А; Левин, М; Лосик, Р. (2004). Молекулярная биология гена (Пятое изд.). Сан-Франциско: Бенджамин Каммингс. ISBN 978-0-321-22368-5.
- ^ а б Майкл В. Пфафф, Алес Тихопад, Кристиан Пргомет и Таня П. Невианс (2005). Определение стабильных генов домашнего хозяйства, дифференциально регулируемых генов-мишеней и целостности образцов: BestKeeper - инструмент на основе Excel с использованием парных корреляций Письма о биотехнологии 26:509–515
- ^ Пфаффл, МВт; Хорган, GW; Демпфл, Л. (2002). «Программный инструмент относительной экспрессии (REST ©) для группового сравнения и статистического анализа результатов относительной экспрессии в ПЦР в реальном времени». Нуклеиновые кислоты Res. 30 (9): e36. Дои:10.1093 / nar / 30.9.e36. ЧВК 113859. PMID 11972351.
- ^ Vandesompele, J; Де Претер, К; Паттин, Ф; Поппе, Б; Ван Рой, N; Де Паэпе, А; Спелеман, Ф (2002). «Точная нормализация количественных данных ОТ-ПЦР в реальном времени путем геометрического усреднения нескольких генов внутреннего контроля». Геномная биология. 3 (7): 1–12. Дои:10.1186 / gb-2002-3-7-research0034. ЧВК 126239. PMID 12184808.
- ^ Рычлик В., Спенсер В. Дж., Роадс Р. Э. (1990). «Оптимизация температуры отжига для амплификации ДНК. in vitro". Нуклеиновые кислоты Res. 18 (21): 6409–6412. Дои:10.1093 / nar / 18.21.6409. ЧВК 332522. PMID 2243783.
- ^ Пфаффл, Майкл (2000). «Разработка и валидация внешне стандартизированной количественной ОТ-ПЦР инсулиноподобного фактора роста-1 с использованием технологии LightCycler SYBR Green I» (PDF). Биохимия (3) - через gene-quantification.org.
- ^ Джозеф Сэмбрук и Дэвид В. Рассел (2001). Молекулярное клонирование: лабораторное руководство (3-е изд.). Колд-Спринг-Харбор, Нью-Йорк: Лаборатория Колд-Спринг-Харбор. ISBN 978-0-87969-576-7.
- ^ Ponchel F; Toomes C; Брансфилд К; Leong F.T; Дуглас С.Х .; Field S.L; Bell S.M; Combaret V; Puisieux A; Мигелл А.Дж. (2003). «ПЦР в реальном времени на основе флуоресценции SYBR-Green I: альтернатива анализу TaqMan для относительной количественной оценки перестроек генов, амплификаций генов и делеций микрогенов». BMC Biotechnol. 3: 18. Дои:10.1186/1472-6750-3-18. ЧВК 270040. PMID 14552656.
- ^ Рири К.М.; Расмуссен Р.П .; Wittwer C.T. (1997). «Дифференциация продуктов с помощью анализа кривых плавления ДНК во время полимеразной цепной реакции» (PDF). Аналитическая биохимия. 245 (2): 154–160. Дои:10.1006 / abio.1996.9916. PMID 9056205.
- ^ Стивен А. Бастин; Владимир Бенеш; Джереми А. Гарсон; Ян Хеллеманс; Джим Хаггетт; Микаэль Кубиста; Рейнхольд Мюллер; Таня Нолан; Майкл В. Пфаффл; Грегори Л. Шипли; Джо Вандесомпеле и Карл Т. Виттвер (апрель 2009 г.). «Рекомендации MIQE: минимум информации для публикации количественных экспериментов ПЦР в реальном времени». Clin. Chem. 55 (4): 611–622. Дои:10.1373 / Clinchem.2008.112797. PMID 19246619.
- ^ а б c Carr, A.C .; Мур, С. Д. (2012). Люсия, Алехандро (ред.). «Надежная количественная оценка полимеразных цепных реакций с использованием глобального фитинга». PLOS ONE. 7 (5): e37640. Bibcode:2012PLoSO ... 737640C. Дои:10.1371 / journal.pone.0037640. ЧВК 3365123. PMID 22701526.
- ^ Schefe JH, Lehmann KE, Buschmann IR, Unger T, Funke-Kaiser H (2006). «Количественный анализ данных ОТ-ПЦР в реальном времени: современные концепции и новая формула« различия CT экспрессии генов »». J Mol Med. 84 (11): 901–910. Дои:10.1007 / s00109-006-0097-6. PMID 16972087.
- ^ Наилис Х., Коэнье Т., Ван Ньивербург Ф., Дефорс Д., Нелис Х. Дж. (2006). «Разработка и оценка различных стратегий нормализации для исследований экспрессии генов в биопленках Candida albicans с помощью ПЦР в реальном времени». BMC Mol. Биол. 7 (1): 25. Дои:10.1186/1471-2199-7-25. ЧВК 1557526. PMID 16889665.
- ^ Нолан Т., Хендс RE, Бастин С.А. (2006). «Количественная оценка мРНК с использованием ОТ-ПЦР в реальном времени». Nat. Protoc. 1 (3): 1559–1582. Дои:10.1038 / nprot.2006.236. PMID 17406449.
- ^ Богги Г., Вульф П.Дж. (2010). Раваси Т. (ред.). «Механистическая модель ПЦР для точной количественной оценки количественных данных ПЦР». PLOS ONE. 5 (8): e12355. Bibcode:2010PLoSO ... 512355B. Дои:10.1371 / journal.pone.0012355. ЧВК 2930010. PMID 20814578.
- ^ Ruijter JM, Pfaffl MW, Zhao S, Spiess AN, Boggy G, Blom J, Rutledge RG, Sisti D, Lievens A, De Preter K, Derveaux S, Hellemans J, Vandesompele J (2012). «Оценка методов анализа кривой КПЦР для надежного обнаружения биомаркеров: систематическая ошибка, разрешение, точность и последствия». Методы. 59 (1): 32–46. Дои:10.1016 / j.ymeth.2012.08.011. PMID 22975077.
- ^ Брюс Гелертер. «PEMF для лечения заболеваний роговицы». lemuriatechnologies.com. Архивировано из оригинал на 2014-06-09.
- ^ Overbergh, L .; Giulietti, A .; Valckx, D .; Decallonne, R .; Bouillon, R .; Матье, К. (2003). «Использование ПЦР с обратной транскриптазой в реальном времени для количественной оценки экспрессии генов цитокинов». Журнал биомолекулярных методов: JBT. 14 (1): 33–43. ЧВК 2279895. PMID 12901609.
- ^ С. Дханасекаран; Т. Марк Доэрти; Джон Кеннет; Исследовательская группа по испытаниям ТБ (март 2010 г.). «Сравнение различных стандартов для абсолютного количественного определения на основе ПЦР в реальном времени». Журнал иммунологических методов. 354 (1–2): 34–39. Дои:10.1016 / j.jim.2010.01.004. PMID 20109462.
- ^ Бар, Цачи; Кубиста, Микаэль; Тихопад, Алесь (19.10.2011). «Подтверждение кинетического сходства в КПЦР». Исследования нуклеиновых кислот. 40 (4): 1395–1406. Дои:10.1093 / nar / gkr778. ISSN 0305-1048. ЧВК 3287174. PMID 22013160.
- ^ Бруннер, AM; Яковлев ИА; Штраус, SH (2004). «Проверка внутреннего контроля для количественных исследований экспрессии генов растений». BMC Plant Biol. 4: 14. Дои:10.1186/1471-2229-4-14. ЧВК 515301. PMID 15317655. Архивировано из оригинал на 2013-08-02.
- ^ МакГеттиган, Пол А (2013). «Транскриптомика в эпоху RNA-seq». Современное мнение в области химической биологии. 17 (1): 4–11. Дои:10.1016 / j.cbpa.2012.12.008. PMID 23290152.
- ^ Теллин, О; Зорзи, В; Lakaye, B; Де Борман, B; Coumans, B; Henne, G; Grisar, T; Игут, А; Хайнен, Э (1999). «Гены домашнего хозяйства как внутренние стандарты: использование и ограничения». J Biotechnol. 75 (2–3): 197–200. Дои:10.1016 / s0168-1656 (99) 00163-7. PMID 10617337.
- ^ Радоник, А; Thulke, S; Маккей, И. М.; Ландт, О; Зигерт, Вт; Ниче, А (2004). «Руководство по выбору референсных генов для количественной ПЦР в реальном времени». Biochem Biophys Res Commun. 313 (4): 856–862. Дои:10.1016 / j.bbrc.2003.11.177. PMID 14706621. Архивировано из оригинал на 2013-08-02.
- ^ Дхеда, К; Huggett, JF; Бустин, С.А.; Джонсон, Массачусетс; Ладья, G; Зумла, А (2004). «Валидация генов домашнего хозяйства для нормализации экспрессии РНК в ПЦР в реальном времени». Биотехнологии. 37 (1): 112–119. Дои:10.2144 / 04371RR03. PMID 15283208.
- ^ Vandesompele J, De Preter K, Pattyn F, Poppe B, Van Roy N, De Paepe A, Speleman F (2002) Точная нормализация количественных данных ОТ-ПЦР в реальном времени путем геометрического усреднения нескольких генов внутреннего контроля " Геном Биол 37: RESEARCH0034
- ^ Пфаффл, МВт; Тихопад, А; Prgomet, C; Невианс, TP (2004). «Определение стабильных генов домашнего хозяйства, дифференциально регулируемых генов-мишеней и целостности образцов: BestKeeper - инструмент на основе Excel с использованием парных корреляций». Biotechnol Lett. 26 (6): 509–515. Дои:10.1023 / б: желчь.0000019559.84305.47. PMID 15127793. Архивировано из оригинал на 2013-08-02.
- ^ Эспи, М.Дж. (январь 2006 г.). «ПЦР в реальном времени в клинической микробиологии: приложения для рутинных лабораторных исследований». Обзоры клинической микробиологии. 19 (3): 165–256. Дои:10.1128 / CMR.19.1.165-256.2006. ЧВК 1360278. PMID 16418529.
- ^ Дхамад, AE; Абдал Рида, Массачусетс (2020). «COVID-19: молекулярные и серологические методы обнаружения». PeerJ. 8: e10180. Дои:10.7717 / peerj.10180. PMID 33083156.
- ^ «Одобренные FDA анализы ОТ-ПЦР и другие молекулярные анализы на вирусы гриппа» (PDF). cdc.gov.
- ^ «rRT-PCR, метод подтверждения случая коронавируса в Ухане - искусственный интеллект для химии». Получено 2020-01-26.
- ^ Филион, М, изд. (2012). Количественная ПЦР в реальном времени в прикладной микробиологии. Caister Academic Press. ISBN 978-1-908230-01-0.
- ^ а б c d е Буше, Блиё, Декьед, Домайзон, Дюфрен, Феррейра, Годон, Хеллаль, Джулиан, Квайзер, Мартин-Лоран, Моффре, Монье, Пейре, Шмитт-Коплин, Сибур, Д'уарон, Биспо, Депортес, Гранд, Куни, Марон, Ранджард (сентябрь 2016 г.). «Методы молекулярной микробиологии для диагностики окружающей среды». Письма по химии окружающей среды. 14 (4): 423–441. Дои:10.1007 / s10311-016-0581-3. Получено 11 мая 2020.CS1 maint: несколько имен: список авторов (связь)
- ^ Болдуин, Б.Г. (1992). «Филогенетическая полезность внутренних транскрибированных спейсеров ядерной рибосомальной ДНК в растениях: пример из Compositaogy». Молекулярная филогенетика и эволюция. 1 (1): 3–16. Дои:10.1016 / 1055-7903 (92) 90030-К. PMID 1342921.
- ^ Томлинсон, Дж. А .; Barker, I .; Бунхэм, Н. (2007). «Более быстрые, простые и более конкретные методы улучшенного молекулярного обнаружения Phytophthora ramorum в полевых условиях». Прикладная и экологическая микробиология. 73 (12): 4040–4047. Дои:10.1128 / AEM.00161-07. ЧВК 1932743. PMID 17449689.
- ^ Холст-Йенсен, Арне; Rønning, Sissel B .; Лёвсет, Астрид; Бердал, Кнут Г. (2003). «Технология ПЦР для скрининга и количественной оценки генетически модифицированных организмов (ГМО)». Аналитическая и биоаналитическая химия. 375 (8): 985–993. Дои:10.1007 / s00216-003-1767-7. PMID 12733008.
- ^ Бродманн П.Д .; Ilg E.C; Berthoud H; Херрманн А. (2002). «… - Методы количественной полимеразной цепной реакции для четырех генетически модифицированных сортов кукурузы…». Журнал AOAC International. 85 (3): 646–653. Дои:10.1093 / jaoac / 85.3.646. PMID 12083257.
- ^ Yeh S.H. Цай С.Ю. Као Дж. Х. Лю C.J. Kuo T.J. Линь М.В. Хуанг В.Л. Лу С.Ф. Джи Дж. Чен Д.С. Другие (2004). «Количественная оценка и генотипирование вируса гепатита В в одной реакции с помощью ПЦР в реальном времени и плавления…». Журнал гепатологии. 41 (4): 659–666. Дои:10.1016 / j.jhep.2004.06.031. PMID 15464248.
- ^ Сотелл Н. М. (1998). «Вероятность реактивации in vivo вируса простого герпеса типа 1 увеличивается с увеличением числа латентно инфицированных нейронов в ганглиях». Журнал вирусологии. 72 (8): 6888–6892. Дои:10.1128 / JVI.72.8.6888-6892.1998. ЧВК 109900. PMID 9658140.
- ^ Питер М. Рости К. Кутюрье Дж. Радвани Ф. Тешима Х. Састре-гарау X. (2006). «Активация MYC, связанная с интеграцией ДНК ВПЧ в локусе MYC в генитальных опухолях». Онкоген. 25 (44): 5985–5993. Дои:10.1038 / sj.onc.1209625. PMID 16682952.
Библиография
- Элиз; Худ, Ален (2002). "La PCR en temps réel: Principes et Applications" (PDF). Обзоры по биологии и биотехнологии. 2 (2): 2–11. Архивировано из оригинал (PDF) на 2009-06-12.
- Бустин, С.А. (2000). «Абсолютное количественное определение мРНК с использованием анализов полимеразной цепной реакции с обратной транскрипцией в реальном времени». Дж Мол Эндокринол. 25 (2): 169–193. Дои:10.1677 / jme.0.0250169. PMID 11013345.
- Higuchi, R .; Dollinger, G .; Walsh, P.S .; Гриффит Р. (1992). «Одновременная амплификация и обнаружение специфических последовательностей ДНК». Биотехнологии. 10 (4): 413–417. Дои:10.1038 / nbt0492-413. PMID 1368485.
- Голландия, P.M .; Abramson, R.D .; Watson, R .; Гельфанд, Д.Х. (1991). «Обнаружение продукта специфической полимеразной цепной реакции с использованием экзонуклеазной активности 50-30 ДНК-полимеразы Thermus aquaticus». Proc. Natl. Акад. Sci. Соединенные Штаты Америки. 88 (16): 7276–7280. Bibcode:1991PNAS ... 88.7276H. Дои:10.1073 / pnas.88.16.7276. JSTOR 2357665. ЧВК 52277. PMID 1871133.
- Кубиста, М; Андраде, JM; Bengtsson, M; Фороотан, А; Jonak, J; Линд, К; Синделка, Р; Sjoback, R; Sjogreen, B; Стромбом, L; Штальберг, А; Зорич, Н. (2006). «Полимеразная цепная реакция в реальном времени». Мол. Аспекты Мед. 27 (2–3): 95–125. Дои:10.1016 / j.mam.2005.12.007. PMID 16460794.
- Higuchi, R .; Fockler, C .; Dollinger, G .; Уотсон, Р. (1993). «Кинетическая ПЦР: мониторинг реакций амплификации ДНК в реальном времени». Биотехнологии. 11 (9): 1026–1030. Дои:10.1038 / nbt0993-1026. PMID 7764001.
- Филион, М. (2012). Количественная ПЦР в реальном времени в прикладной микробиологии. Caister Academic Press. ISBN 978-1-908230-01-0.
- Wawrik, B; Пол, JH; Табита, FR (2002). "Количественная оценка ПЦР в реальном времени rbcL (рибулозо-1,5-бисфосфаткарбоксилаза / оксигеназа) мРНК у диатомовых водорослей и пелагофитов ». Appl. Environ. Микробиол. 68 (8): 3771–3779. Дои:10.1128 / aem.68.8.3771-3779.2002. ЧВК 123995. PMID 12147471.
- Логан Дж; Эдвардс К; Сондерс Н, ред. (2009). ПЦР в реальном времени: современные технологии и приложения. Caister Academic Press. ISBN 978-1-904455-39-4.
