Сплайсосома - Spliceosome
А сплайсосома большой и сложный молекулярная идентичность РНК находится в основном в ядро из эукариотические клетки. Сплайсосома собирается из малые ядерные РНК (мяРНК ) и примерно 80 белков. Сплайсосома удаляет интроны из записано пре-мРНК, тип первичная стенограмма. Этот процесс обычно называют сращивание.[1] Аналогия - редактор фильма, который выборочно вырезает нерелевантный или неправильный материал (эквивалент интроны ) из исходного фильма и отправляет исправленную версию режиссеру для окончательной сборки.

История
В 1977 г. работа Острый и Робертс лаборатории показали, что гены высших организмов «расщеплены» или присутствуют в нескольких отдельных сегментах молекулы ДНК.[2][3] Кодирующие области гена разделены некодирующей ДНК, которая не участвует в экспрессии белка. Структура расщепленного гена была обнаружена при гибридизации аденовирусных мРНК с фрагментами одноцепочечной вирусной ДНК, расщепляемыми эндонуклеазой.[2] Было обнаружено, что мРНК гибридов мРНК-ДНК содержат 5' и 3' хвосты областей, не связанных водородными связями. Когда использовались более крупные фрагменты вирусной ДНК, при гибридизации с вирусными мРНК наблюдались разветвленные структуры петлевой ДНК. Выяснилось, что закольцованные области, интроны, вырезаются из мРНК-предшественников в процессе, который Шарп назвал «сплайсинг». Впоследствии было установлено, что структура расщепленного гена является общей для эукариотический гены. Филипп Шарп и Ричард Дж. Робертс были награждены 1993 Нобелевская премия по физиологии и медицине за открытие интронов и процесса сплайсинга.
Сочинение
Каждая сплайсосома состоит из пяти малые ядерные РНК (мяРНК) и ряд связанных белковых факторов. Когда эти малые РНК объединяются с белковыми факторами, они образуют комплексы РНК-белок, называемые snRNPs (sторговый центр пнеясный рИбопucleo пroteins, произносится как snurps). мяРНК, которые составляют главную сплайсосому, называются U1, U2, U4, U5, и U6, так называемые, потому что они богаты уридин, и участвуют в нескольких взаимодействиях РНК-РНК и РНК-белок.[1]
Каноническая сборка сплайсосомы происходит заново на каждом пре-мРНК (также известна как гетерогенная ядерная РНК). Пре-мРНК содержит определенные элементы последовательности, которые распознаются и используются во время сборки сплайсосом. К ним относятся 5'-концевой сайт сплайсинга, последовательность точки ветвления, полипиримидиновый тракт и 3'-концевой сайт сплайсинга. Сплайсосома катализирует удаление интронов и лигирование фланкирующих экзонов.
Интроны обычно имеют нуклеотидную последовательность GU на 5'-конце сайта сплайсинга и AG на 3'-концевом сайте сплайсинга. 3'-сайт сплайсинга может быть дополнительно определен полипиримидином переменной длины, называемым полипиримидиновый тракт (PPT), который выполняет двойную функцию: рекрутируя факторы в 3'-сайт сплайсинга и, возможно, рекрутируя факторы в последовательность точек ветвления (БПС). BPS содержит сохраненные аденозин требуется для первого этапа сварки.
Многие белки обладают мотивом, связывающим цинк, что подчеркивает важность цинка в механизме сплайсинга.[4][5][6] О первой реконструкции с молекулярным разрешением тройного комплекса малых ядерных рибонуклеопротеидов U4 / U6.U5 (три-мяРНП) было сообщено в 2016 году.[7]
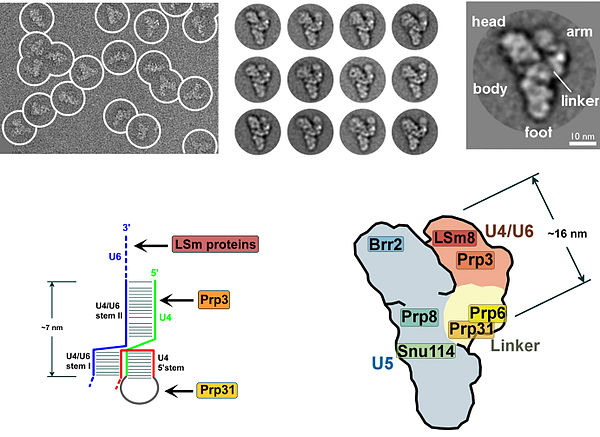
Крио-ЭМ широко применялась Ши и соавторами для выяснения почти атомной структуры сплайсосом в обоих дрожжевых грибах.[9] и люди.[10] Молекулярный каркас сплайсосомы с разрешением, близким к атомному, демонстрирует, что компонент Spp42 U5 snRNP образует центральный каркас и заякоривает каталитический центр у дрожжей. Атомная структура сплайсосомы человека иллюстрирует, что компонент стадии II Slu7 принимает расширенную структуру, готовую к выбору сайта 3'-сплайсинга. Все пять металлов (обозначенных как Mg2 +) в дрожжевом комплексе сохраняются в человеческом комплексе.
Альтернативная сварка
Альтернативная сварка (повторное совмещение разных экзоны ) является основным источником генетическое разнообразие у эукариот. Варианты сращивания были использованы для учета относительно небольшого количества белков, кодирующих гены в человеческий геном, в настоящее время оценивается примерно в 20 000 экземпляров. Один конкретный Дрозофила ген, Dscam, предполагалось, что они будут альтернативно объединены в 38000 различных мРНК, предполагая, что все его экзоны могут сплавляться независимо друг от друга.[11]
сборка
Модель формирования активного сайта сплайсосомы включает упорядоченную, ступенчатую сборку дискретных частиц snRNP на субстрате пре-мРНК. Первое распознавание пре-мРНК включает связывание U1 snRNP с 5'-концом сайта сплайсинга пре-мРНК и другими факторами, не связанными с snRNP, с образованием коммитирующего комплекса или раннего (E) комплекса у млекопитающих.[12][13] Коммитирующий комплекс представляет собой АТФ-независимый комплекс, который связывает пре-мРНК с путем сплайсинга.[14] U2 snRNP рекрутируется в область разветвления через взаимодействия с компонентом комплекса E U2AF (Вспомогательный фактор U2 snRNP) и, возможно, U1 snRNP. В АТФ-зависимой реакции U2 snRNP становится тесно связанным с последовательностью точки ветвления (BPS) с образованием комплекса A. Дуплекс, образованный между U2 snRNP и областью разветвления пре-мРНК, выпячивается из ответвления аденозина, определяя его как нуклеофил для первая переэтерификация.[15]
Наличие псевдоуридин остаток в U2 мяРНК, почти противоположный сайту разветвления, приводит к измененной конформации дуплекса РНК-РНК при связывании U2 мяРНП. В частности, измененная структура дуплекса, вызванная псевдоуридином, помещает 2'-ОН выпуклого аденозина в благоприятное положение для первой стадии сплайсинга.[16] U4 / U5 / U6 tri-snRNP (см. Рисунок 1) рекрутируется в собирающуюся сплайсосому с образованием комплекса B, и после нескольких перестроек комплекс C активируется для катализа.[17][18] Неясно, как tri-snRNP рекрутируется в комплекс A, но этот процесс может быть опосредован посредством белок-белковых взаимодействий и / или взаимодействий спаривания оснований между U2 snRNA и U6 snRNA.
U5 snRNP взаимодействует с последовательностями в сайтах сплайсинга 5 'и 3' через инвариантную петлю мяРНК U5[19] и компоненты белка U5 взаимодействуют с 3'-участком сплайсинга.[20]
При рекрутировании три-мяРНП несколько перегруппировок РНК-РНК предшествуют первой каталитической стадии, и дальнейшие перегруппировки происходят в каталитически активной сплайсосоме. Некоторые из взаимодействий РНК-РНК исключают друг друга; однако неизвестно, что запускает эти взаимодействия, ни порядок этих перестроек. Первая перестройка - это, вероятно, смещение U1 snRNP из 5'-сайта сплайсинга и образование взаимодействия U6 snRNA. Известно, что U1 snRNP слабо связан с полностью сформированными сплайсосомами,[21] и U1 snRNP ингибирует образование взаимодействия сайта сплайсинга U6-5 'на модели субстратного олигонуклеотида, содержащего короткий 5'-экзон и 5'-сайт сплайсинга.[22] Связывание U2 snRNP с последовательностью точки ветвления (BPS) является одним из примеров взаимодействия РНК-РНК, замещающего взаимодействие белок-РНК. При привлечении U2 snRNP, ответвление связывающего белка SF1 в коммитерном комплексе смещается, поскольку сайт связывания U2 snRNA и SF1 являются взаимоисключающими событиями.
Внутри U2 snRNA существуют другие взаимоисключающие перестройки, которые происходят между конкурирующими конформациями. Например, в активной форме предпочтительна петля ствола IIa; в неактивной форме преобладает взаимоисключающее взаимодействие между петлей и последующей последовательностью.[18] Неясно, как U4 вытесняется из U6 snRNA, хотя РНК участвует в сборке сплайсосом и может действовать, чтобы раскручивать U4 / U6 и способствовать образованию взаимодействия U2 / U6 snRNA. Взаимодействия петель стебля I и II U4 / U6 диссоциируют, и освобожденная область петли стебля II U6 складывается сама на себя с образованием внутримолекулярной петли стебля, и U4 больше не требуется для дальнейшей сборки сплайсосом. Освобожденная область петли стебля I пар оснований U6 с мяРНК U2, образующая спираль I U2 / U6. Однако структура спирали I является взаимоисключающей с 3'-половиной внутренней 5'-области петли стебля мяРНК U2.
Незначительная сплайсосома
Немного эукариоты имеют вторую сплайсосому, так называемую второстепенная сплайсосома.[23]Группа менее распространенных мяРНК, U11, U12, U4atac, и U6atac вместе с U5 представляют собой субъединицы минорной сплайсосомы, которая сплайсирует редкий класс пре-мРНК интронов, обозначенный как U12-тип. Минорная сплайсосома расположена в ядре, как и ее главный двойник,[24] хотя есть исключения в некоторых специализированных клетках, включая безъядерные тромбоциты[25] и дендроплазма (дендрит цитоплазма) нейрональных клеток.[26]
Рекомендации
- ^ а б Will CL, Lührmann R (июль 2011 г.). «Структура и функция сплайсосом». Перспективы Колд-Спринг-Харбор в биологии. 3 (7): a003707. Дои:10.1101 / cshperspect.a003707. ЧВК 3119917. PMID 21441581.
- ^ а б Бергет С.М., Мур С., Шарп П.А. (август 1977 г.). «Сплайсированные сегменты на 5'-конце поздней мРНК аденовируса 2». Труды Национальной академии наук Соединенных Штатов Америки. 74 (8): 3171–5. Bibcode:1977ПНАС ... 74.3171Б. Дои:10.1073 / pnas.74.8.3171. ЧВК 431482. PMID 269380.
- ^ Чоу LT, Робертс Дж. М., Льюис Дж. Б., Брокер Т. Р. (август 1977 г.). «Карта цитоплазматических транскриптов РНК литического аденовируса типа 2, определенная с помощью электронной микроскопии гибридов РНК: ДНК». Клетка. 11 (4): 819–36. Дои:10.1016 / 0092-8674 (77) 90294-Х. PMID 890740.
- ^ Агафонов Д.Е., Декерт Дж., Вольф Э., Оденвельдер П., Бессонов С., Уилл К.Л., Урлауб Х., Люрманн Р. (июль 2011 г.). «Полуколичественный протеомный анализ сплайсосомы человека с помощью нового метода двумерного гель-электрофореза». Молекулярная и клеточная биология. 31 (13): 2667–82. Дои:10.1128 / mcb.05266-11. ЧВК 3133382. PMID 21536652.
- ^ Кун А.Н., ван Сантен М.А., Швинхорст А., Урлауб Н., Люрманн Р. (январь 2009 г.). «Замедление сборки сплайсосом на различных стадиях низкомолекулярными ингибиторами ацетилирования и деацетилирования белков». РНК. 15 (1): 153–75. Дои:10.1261 / rna.1332609. ЧВК 2612777. PMID 19029308.
- ^ Патил В., Канцонери Дж. К., Саматов Т. Р., Люрманн Р., Ойелере А. К. (сентябрь 2012 г.). «Молекулярная архитектура малых молекул, хелатирующих цинк, которые ингибируют сборку сплайсосом на ранней стадии». РНК. 18 (9): 1605–11. Дои:10.1261 / rna.034819.112. ЧВК 3425776. PMID 22832025.
- ^ Кейт Дж. Х. (март 2016 г.). «СТРУКТУРА. Большой взрыв в структурной биологии сплайсосом». Наука. 351 (6280): 1390–2. Дои:10.1126 / science.aaf4465. PMID 27013712.
- ^ Хекер И., Сандер Б., Голас М.М., Вольф Е., Карагез Е., Кастнер Б., Старк Н., Фабрицио П., Люрманн Р. (ноябрь 2008 г.). «Локализация белков Prp8, Brr2, Snu114 и U4 / U6 в три-snRNP дрожжей с помощью электронной микроскопии». Структурная и молекулярная биология природы. 15 (11): 1206–12. Дои:10.1038 / nsmb.1506. PMID 18953335.
- ^ Ян Ц., Ханг Дж., Ван Р., Хуанг М., Вонг С.К., Ши И (сентябрь 2015 г.). «Структура сплайсосомы дрожжей при разрешении 3,6 ангстрем». Наука. 349 (6253): 1182–91. Bibcode:2015Научный ... 349.1182Y. Дои:10.1126 / science.aac7629. PMID 26292707.
- ^ Чжан X, Ян Ц., Ханг Дж., Финчи Ли, Лэй Дж, Ши И (май 2017 г.). «Атомная структура сплайсосомы человека». Клетка. 169 (5): 918–929.e14. Дои:10.1016 / j.cell.2017.04.033. PMID 28502770.
- ^ Schmucker D, Clemens JC, Shu H, Worby CA, Xiao J, Muda M, Dixon JE, Zipursky SL (июнь 2000 г.). «Drosophila Dscam - это рецептор управления аксоном, демонстрирующий необычайное молекулярное разнообразие». Клетка. 101 (6): 671–84. Дои:10.1016 / S0092-8674 (00) 80878-8. PMID 10892653.
- ^ Джеймисон С.Ф., Ворон А., Гарсиа-Бланко М.А. (октябрь 1992 г.). «Путь сборки сплайсосом в экстрактах млекопитающих». Молекулярная и клеточная биология. 12 (10): 4279–87. Дои:10.1128 / MCB.12.10.4279. ЧВК 360351. PMID 1383687.
- ^ Серафин Б, Росбаш М (октябрь 1989 г.). «Идентификация функциональных комплексов мяРНК U1-пре-мРНК, преданных сборке и сплайсингу сплайсосом». Клетка. 59 (2): 349–58. Дои:10.1016/0092-8674(89)90296-1. PMID 2529976.
- ^ Легрен П., Серафин Б., Росбаш М. (сентябрь 1988 г.). «Ранняя приверженность дрожжевой пре-мРНК к сплайсосомному пути». Молекулярная и клеточная биология. 8 (9): 3755–60. Дои:10.1128 / MCB.8.9.3755. ЧВК 365433. PMID 3065622.
- ^ Query CC, Мур MJ, Sharp PA (март 1994). «Отбор нуклеофилов ответвлений в сплайсинге пре-мРНК: доказательства модели выпуклого дуплекса». Гены и развитие. 8 (5): 587–97. Дои:10.1101 / gad.8.5.587. PMID 7926752.
- ^ Ньюби М. И., Гринбаум Н. Л. (декабрь 2002 г.). «Формирование мотива распознавания сайта сплайсосомной ветви с помощью консервативного псевдоуридина». Структурная биология природы. 9 (12): 958–65. Дои:10.1038 / nsb873. PMID 12426583.
- ^ Burge CB, et al. (1999). «Сплайсинг предшественников мРНК сплайсосомами». В Gesteland RF, Чех Т.Р., Аткинс Дж.Ф. (ред.). Мир РНК. Лаборатория Колд-Спринг-Харбор. Нажмите. С. 525–60. ISBN 978-0-87969-380-0.
- ^ а б Стейли Дж. П., Гатри С. (февраль 1998 г.). «Механические устройства сплайсосомы: моторы, часы, пружины и прочее». Клетка. 92 (3): 315–26. Дои:10.1016 / S0092-8674 (00) 80925-3. PMID 9476892.
- ^ Ньюман AJ, Teigelkamp S, Beggs JD (ноябрь 1995 г.). «Взаимодействие мяРНК в 5 'и 3'-сайтах сплайсинга отслеживается с помощью фотоактивированного перекрестного связывания в сплайсосомах дрожжей». РНК. 1 (9): 968–80. ЧВК 1369345. PMID 8548661.
- ^ Кьяра, доктор медицины, Паланджян Л., Фельд Крамер Р., Рид Р. (август 1997 г.). «Доказательства того, что U5 snRNP распознает 3'-сайт сплайсинга для каталитической стадии II у млекопитающих». Журнал EMBO. 16 (15): 4746–59. Дои:10.1093 / emboj / 16.15.4746. ЧВК 1170101. PMID 9303319.
- ^ Мур MJ, Sharp PA (сентябрь 1993 г.). «Доказательства наличия двух активных сайтов в сплайсосоме, полученные с помощью стереохимии сплайсинга пре-мРНК». Природа. 365 (6444): 364–8. Bibcode:1993Натура.365..364М. Дои:10.1038 / 365364a0. PMID 8397340.
- ^ Konforti BB, Koziolkiewicz MJ, Konarska MM (декабрь 1993 г.). «Нарушение спаривания оснований между 5'-сайтом сплайсинга и 5'-концом U1 мяРНК требуется для сборки сплайсосомы». Клетка. 75 (5): 863–73. Дои:10.1016 / 0092-8674 (93) 90531-Т. PMID 8252623.
- ^ Патель А.А., Steitz JA (Декабрь 2003 г.). «Двойной сплайсинг: выводы из второй сплайсосомы». Обзоры природы. Молекулярная клеточная биология. 4 (12): 960–70. Дои:10.1038 / nrm1259. PMID 14685174.
- ^ Песса Х.К., Уилл К.Л., Менг Х, Шнайдер С., Уоткинс, штат Нью-Джерси, Перэля Н., Нимарк М., Турунен Дж. Дж., Люрманн Р., Фриландер М. Дж. (Июнь 2008 г.). «Незначительные компоненты сплайсосом преимущественно локализуются в ядре». Труды Национальной академии наук Соединенных Штатов Америки. 105 (25): 8655–60. Дои:10.1073 / pnas.0803646105. ЧВК 2438382. PMID 18559850.
- ^ Денис М.М., Толли Н.Д., Бантинг М., Шверц Х., Цзян Х., Линдеманн С., Йост С.К., Рубнер Ф.Дж., Альбертина К.Х., Свобода К.Дж., Фратто С.М., Толли Э., Крайсс Л.В., Макинтайр TM, Циммерман Г.А., Вейрих А.С. (август 2005 г. ). «Выход за пределы ядра: зависимый от сигнала сплайсинг пре-мРНК в безъядерных тромбоцитах». Клетка. 122 (3): 379–91. Дои:10.1016 / j.cell.2005.06.015. ЧВК 4401993. PMID 16096058.
- ^ Гланзер Дж., Мияширо К.Ю., Сул Дж.Й., Барретт Л., Бельт Б, Хейдон П., Эбервин Дж. (Ноябрь 2005 г.). «Возможность сплайсинга РНК живых дендритов нейронов». Труды Национальной академии наук Соединенных Штатов Америки. 102 (46): 16859–64. Bibcode:2005PNAS..10216859G. Дои:10.1073 / pnas.0503783102. ЧВК 1277967. PMID 16275927.
дальнейшее чтение
- Мясник SE (2011). «Глава 8. Сплайсосома и ее ионы металлов». В Sigel A, Sigel H, Sigel RK (ред.). Структурная и каталитическая роль ионов металлов в РНК. Ионы металлов в науках о жизни. 9. Издательство РСК. С. 235–51. Дои:10.1039/9781849732512-00235. ISBN 978-1-84973-094-5.
- Нильсен Т.В. (декабрь 2003 г.). «Сплайсосома: самая сложная макромолекулярная машина в клетке?». BioEssays. 25 (12): 1147–9. Дои:10.1002 / bies.10394. PMID 14635248.
внешняя ссылка
- Трехмерные макромолекулярные структуры сплайсосом из банка данных EM (EMDB)
- Сплайсосомы в Национальной медицинской библиотеке США Рубрики медицинской тематики (MeSH)
